Le glycogène et l’amidon, polymères de glucose de taille mégadaltonique, constituent le principal réservoir d’énergie et de composés carbonés facilement disponibles dans la plupart des organismes vivants. Chez les mammifères, l’absorption et l’utilisation du glucose sont étroitement contrôlées. Des défauts dans la manipulation normale du glucose sont associés à une variété de pathologies humaines telles que les maladies de stockage du glycogène et le diabète, dans lequel une hyperglycémie persistante est corrélée à un début précoce et à une gravité accrue de la maladie. Les voies métaboliques de la conversion catalytique du glycogène font intervenir plusieurs enzymes, dont la glycogène synthase qui catalyse l’élongation et la glycogène phosphorylase qui décompose les glucanes a(1,4)-liés. Les études biochimiques et structurelles des glycogène et maltodextrine phosphorylases au cours des 30 dernières années ont permis d’acquérir une grande quantité d’informations sur la dégradation du glycogène. Ces études ont montré que la régulation de la glycogène phosphorylase est un exemple fascinant d’allostérie covalente et non covalente, coordonnée de manière exquise par le contrôle hormonal. Des informations décrivant les structures tertiaires et quaternaires sont maintenant disponibles pour pratiquement toutes les enzymes directement impliquées dans la transformation du glycogène. De manière surprenante, très peu d’informations structurelles ont été rapportées sur la glycogène synthase, qui catalyse l’allongement du glycogène par l’addition successive de résidus de glucose ![]() -(1,4)-liés à l’extrémité non réductrice du polymère, en utilisant l’ADP/UDP-glucose comme donneur de glucose.
-(1,4)-liés à l’extrémité non réductrice du polymère, en utilisant l’ADP/UDP-glucose comme donneur de glucose.
Nous avons maintenant déterminé la structure cristalline de la glycogène synthase d’Agrobacterium tumefaciens (AtGS) à une résolution de 2,3 Å, à la fois dans sa forme apo et en présence d’ADP. Les données ont été recueillies sur ID29, et la structure a été résolue en utilisant la technique SAD (diffraction anormale à une seule longueur d’onde), suivie de procédures de modification de la densité. La protéine se présente sous la forme d’un dimère dans l’unité asymétrique, chaque monomère étant composé de deux domaines ‘Rossmann-fold’ (Figure 81) qui sont organisés comme dans les structures de la glycogène phosphorylase et d’autres glycosyltransférases de la superfamille GT-B . Une fissure profonde entre les deux domaines contient le centre catalytique. Une différence majeure entre les glycogènes synthases des mammifères (famille GT3) et les glycogènes synthases bactériennes (GT5), est que seules les enzymes GT3 sont étroitement régulées par la phosphorylation et la modulation allostérique non covalente. Selon les alignements de séquences multiples basés sur la structure entre les séquences GT3, GT5 et glycogène phosphorylase, les sites de phosphorylation régulateurs dans la glycogène synthase humaine sont probablement situés dans les insertions N-terminales de 25 résidus et C-terminales de 120 résidus, en dehors du noyau GT-B non régulé des glycogène synthases bactériennes. Des études de mutagenèse avaient précédemment identifié deux groupes Arg conservés dans les enzymes GT3 comme faisant partie du ou des sites allostériques pour les résidus phospho-Ser/Thr et l’activateur non covalent Glc-6P. Les résidus équivalents dans l’AtGS sont situés à une extrémité du domaine C-terminal, loin du centre de réaction, indiquant un effet régulateur à longue distance.
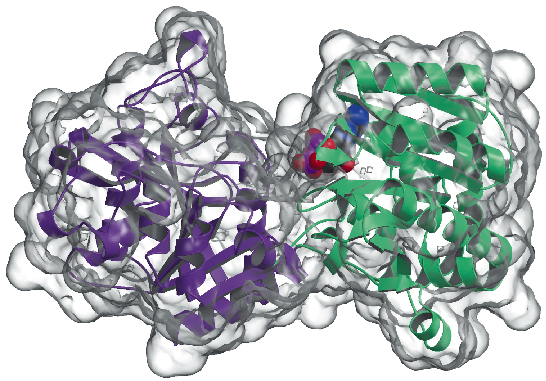
Fig. 81 : Structure de la glycogène synthase : la surface accessible au solvant est représentée en transparence. Notez la molécule d’ADP liée (dessinée sous forme de boules VDW).
La structure de l’AtGS présente une ressemblance topologique frappante avec les structures centrales des glycogène/maltodextrine phosphorylases (famille GT35) (Figure 82a), même si les identités de séquence sont faibles (~14%). La structure de chaque domaine de Rossmann de l’AtGS est significativement plus proche de celles des phosphorylases de la famille GT35 que de toute autre glycosyltransférase nucléotide-dépendante. La glycogène synthase et les phosphorylases présentent également une forte ressemblance au niveau de leurs sites catalytiques (Figure 82b). Le ribose ADP et les groupes pyridoxal se trouvent dans des positions équivalentes, tout comme le phosphate distal de l’ADP dans l’AtGS et le substrat de phosphate inorganique dans les phosphorylases. En outre, les acides aminés critiques qui interagissent avec les parties glucose des substrats sont également conservés. Le mécanisme catalytique utilisé par les glycogène synthases et les phosphorylases est donc probablement similaire, des changements subtils dans la structure expliquant leurs propriétés fonctionnelles globalement différentes.
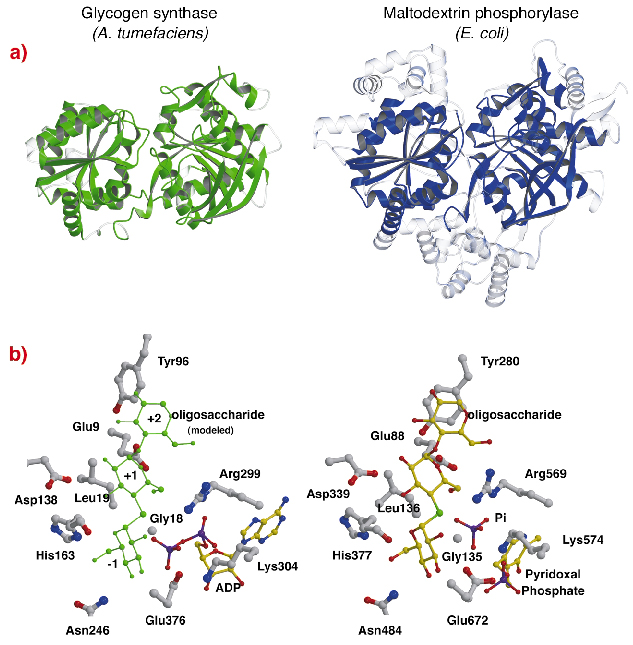
Fig. 82 : Homologie avec la glycogène phosphorylase : (a) après alignement structural, les résidus équivalents superposés entre l’AtGS et la maltodextrine phosphorylase MalP d’E. coli, sont représentés en aplat (régions non alignées, en transparent) ; (b) les sites catalytiques de l’AtGS et de la MalP.
La suggestion initiale que la synthèse du glycogène représentait simplement une inversion de sa phosphorolyse dégradative a été démontrée comme étant erronée lorsque Leloir a montré que l’UDP-Glc et la glycogène synthase, et non le Glc-1-P et la glycogène phosphorylase, étaient responsables de l’allongement des polysaccharides . Nos travaux démontrent cependant maintenant que les réactions indépendantes opposées dans l’interconversion glucose-glycogène sont catalysées par des enzymes homologues étroitement liées.