Isocitratdehydrogenase katalysiert die chemischen Reaktionen:
Isocitrat + NAD+ ⇌ {\displaystyle \rightleftharpoons }

2-Oxoglutarat + CO2 + NADH + H+ Isocitrat + NADP+ ⇌ {\displaystyle \rightleftharpoons }

2-Oxoglutarat + CO2 + NADPH + H+
Die freie Gesamtenergie für diese Reaktion beträgt -8,4 kJ/mol.
SchritteBearbeiten
Im Rahmen des Zitronensäurezyklus wird Isocitrat, das aus der Isomerisierung von Citrat entsteht, sowohl oxidiert als auch decarboxyliert. Mit Hilfe des Enzyms Isocitrat-Dehydrogenase (IDH) wird Isocitrat in seinem aktiven Zentrum von den umgebenden Aminosäuren Arginin, Tyrosin, Asparagin, Serin, Threonin und Asparaginsäure gehalten. Der erste Kasten zeigt die Gesamtreaktion der Isocitrat-Dehydrogenase. Die für das Funktionieren dieses Enzymmechanismus erforderlichen Reaktanten sind Isocitrat, NAD+/NADP+ und Mn2+ oder Mg2+. Die Produkte der Reaktion sind Alpha-Ketoglutarat, Kohlendioxid und NADH + H+/NADPH + H+. Wassermoleküle werden zur Deprotonierung der Sauerstoffatome (O3) von Isocitrat verwendet.
Das zweite Feld ist Schritt 1, die Oxidation des alpha-C (C#2). Die Oxidation ist der erste Schritt, den Isocitrat durchläuft. Dabei wird die Alkoholgruppe am Alpha-Kohlenstoff (C#2) deprotoniert, und die Elektronen fließen zum Alpha-C, wobei eine Ketongruppe gebildet und ein Hydrid von C#2 entfernt wird, wobei NAD+/NADP+ als elektronenaufnehmender Cofaktor verwendet wird. Die Oxidation des alpha-C ermöglicht eine Position, in der Elektronen (im nächsten Schritt) von der Carboxylgruppe nach unten kommen und die Elektronen (die den doppelt gebundenen Sauerstoff bilden) wieder nach oben auf den Sauerstoff drücken oder ein Proton von einer nahegelegenen Lysin-Aminosäure aufnehmen.
Das dritte Feld ist Schritt 2, die Decarboxylierung von Oxalosuccinat. In diesem Schritt wird der Sauerstoff der Carboxylgruppe durch eine nahegelegene Tyrosin-Aminosäure deprotoniert, und diese Elektronen fließen zum Kohlenstoff 2 hinunter. Das Kohlendioxid verlässt den Beta-Kohlenstoff des Isocitrats als Abgangsgruppe, wobei die Elektronen zum Ketonsauerstoff des Alpha-C fließen und eine negative Ladung auf den Sauerstoff des Alpha-C legen und eine ungesättigte Alpha-Beta-Doppelbindung zwischen den Kohlenstoffen 2 und 3 bilden. Das einsame Paar am alpha-C-Sauerstoff nimmt ein Proton von einer nahegelegenen Lysin-Aminosäure auf.
Das vierte Feld ist Schritt 3, der die Sättigung der ungesättigten alpha-beta-Doppelbindung zwischen den Kohlenstoffen 2 und 3 darstellt. In diesem Reaktionsschritt deprotoniert Lysin den Sauerstoff des alpha-Kohlenstoffs und das einsame Elektronenpaar am Sauerstoff des alpha-Kohlenstoffs kommt herunter, reformiert die Keton-Doppelbindung und stößt das einsame Elektronenpaar (das die Doppelbindung zwischen dem alpha- und beta-Kohlenstoff bildet) ab, wobei es ein Proton von der benachbarten Tyrosin-Aminosäure aufnimmt. Diese Reaktion führt zur Bildung von alpha-Ketoglutarat, NADH + H+/NADPH + H+ und CO2.
Detaillierter MechanismusBearbeiten
Zwei Aspartat-Aminosäurereste (unten links) stehen in Wechselwirkung mit zwei benachbarten Wassermolekülen (w6 und w8) im Mn2+-Isocitrat-Schweine-IDH-Komplex, um den Alkohol vom alpha-Kohlenstoffatom zu deprotonieren. Die Oxidation des alpha-C-Atoms findet ebenfalls in diesem Bild statt, wobei NAD+ ein Hydrid aufnimmt und Oxalosuccinat bildet. Zusammen mit der stereochemischen Veränderung von sp3 zu sp2 um das alpha-C herum wird eine Ketongruppe aus der Alkoholgruppe gebildet. Die Bildung dieser Keton-Doppelbindung ermöglicht eine Resonanz, da sich die Elektronen von der abgehenden Carboxylatgruppe zum Keton bewegen.
Die Decarboxylierung von Oxalosuccinat (unten in der Mitte) ist ein wichtiger Schritt bei der Bildung von alpha-Ketoglutarat. Bei dieser Reaktion abstrahiert das einsame Paar am benachbarten Tyrosin-Hydroxyl das Proton von der Carboxylgruppe. Diese Carboxylgruppe wird auch als Beta-Untereinheit des Isocitratmoleküls bezeichnet. Die Deprotonierung der Carboxylgruppe bewirkt, dass sich das einsame Elektronenpaar nach unten bewegt und Kohlendioxid bildet und sich vom Oxalosuccinat trennt. Die Elektronen bewegen sich weiter in Richtung des Alpha-Kohlenstoffs und schieben die Elektronen der Doppelbindung (die das Keton bilden) nach oben, um ein Proton von einem benachbarten Lysinrest zu entziehen. Es entsteht eine ungesättigte Alpha-Beta-Doppelbindung zwischen Kohlenstoff 2 und drei. Wie Sie in der Abbildung sehen können, stellt das grüne Ion entweder Mg2+ oder Mn2+ dar, das als Cofaktor für diese Reaktion erforderlich ist. Das Metall-Ion bildet einen kleinen Komplex durch ionische Wechselwirkungen mit den Sauerstoffatomen am vierten und fünften Kohlenstoff (auch bekannt als die Gamma-Untereinheit von Isocitrat).
Nachdem das Kohlendioxid im Decarboxylierungsschritt (unten rechts) vom Oxalosuccinat abgespalten wurde, tautomerisiert das Enol zum Keto. Die Bildung der Keton-Doppelbindung wird durch die Deprotonierung des Sauerstoffs am Alpha-Kohlenstoff (C#2) durch dasselbe Lysin eingeleitet, das den Sauerstoff zuerst protoniert hat. Das einsame Elektronenpaar wandert nach unten und stößt dabei die einsamen Elektronenpaare ab, die die Doppelbindung gebildet haben. Dieses einsame Elektronenpaar abstrahiert ein Proton aus dem Tyrosin, das die Carboxylgruppe im Decarboxylierungsschritt deprotoniert hat. Der Grund, warum wir sagen können, dass die Lys- und Tyr-Reste die gleichen sind wie im vorherigen Schritt, ist, dass sie dabei helfen, das Isocitratmolekül im aktiven Zentrum des Enzyms zu halten. Diese beiden Reste sind in der Lage, Wasserstoffbrückenbindungen hin und her zu bilden, solange sie nahe genug am Substrat sind.
 Oxidoreduktaseschritt, bei dem NAD+ zur Aufnahme eines Hydrids verwendet wird.
|
 Decarboxylierung von Oxalosuccinat.
|
 Sättigung der alpha-beta-ungesättigten Doppelbindung.
|
Das Isocitrat-Dehydrogenase-Enzym produziert, wie oben beschrieben, Alpha-Ketoglutarat, Kohlendioxid und NADH + H+/NADPH + H+. Bei der Reaktion finden drei Veränderungen statt. Die Oxidation von Kohlenstoff 2, die Decarboxylierung (Verlust von Kohlendioxid) von Kohlenstoff 3 und die Bildung einer Ketongruppe mit einer stereochemischen Änderung von sp3 zu sp2.
 |
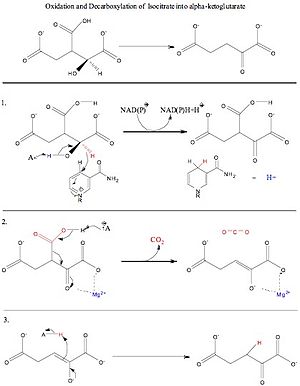
 Porcine mitochondriale NADP+-abhängige Isocitrat-Dehydrogenase im Komplex mit Mn2+ und Isocitrat. Oberflächenansicht der Tasche des aktiven Zentrums, in der Isocitrat durch polare Aminosäuren gebunden ist.
|
 Porcine mitochondriale NADP+-abhängige Isocitrat-Dehydrogenase im Komplex mit Mn2+ und Isocitrat.
|
 Schweinischer Enzymkomplex; aktives Zentrum Isocitrat und angrenzendes A.A.
|
Aktives ZentrumBearbeiten
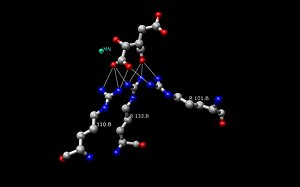
Die Struktur des Enzyms Isocitrat-Dehydrogenase (IDH) in Escherichia coli war die erste Struktur, die aufgeklärt und verstanden wurde. Seitdem wird die IDH-Struktur von Escherichia coli von den meisten Forschern verwendet, um Vergleiche mit anderen Isocitrat-Dehydrogenase-Enzymen anzustellen. Über dieses bakterielle Enzym ist viel Detailwissen vorhanden, und es hat sich gezeigt, dass die meisten Isocitrat-Dehydrogenasen in ihrer Struktur und damit auch in ihrer Funktion ähnlich sind. Diese Ähnlichkeit von Struktur und Funktion legt die Vermutung nahe, dass die Strukturen ebenso wie die Aminosäuren konserviert sind. Daher sollten auch die aktiven Stellen der meisten prokaryotischen Isocitrat-Dehydrogenase-Enzyme konserviert sein, was in vielen Studien über prokaryotische Enzyme beobachtet wurde. Die eukaryotischen Isocitrat-Dehydrogenase-Enzyme hingegen sind noch nicht vollständig erforscht: Jedes IDH-Dimer hat zwei aktive Stellen. Jede aktive Stelle bindet ein NAD+/NADP+-Molekül und ein zweiwertiges Metallion (Mg2+, Mn2+). Im Allgemeinen hat jede aktive Stelle eine konservierte Aminosäuresequenz für jede spezifische Bindungsstelle. In Desulfotalea psychrophila (DpIDH) und Schwein (PcIDH) sind drei Substrate an die aktive Stelle gebunden.
- Isocitrat bindet innerhalb der aktiven Stelle an eine konservierte Sequenz von etwa acht Aminosäuren durch Wasserstoffbrücken. Zu diesen Säuren gehören (die Rückstände können variieren, haben aber ähnliche Eigenschaften) Tyrosin, Serin, Asparagin, Arginin, Arginin, Arginin, Tyrosin und Lysin. Ihre Positionen auf dem Rückgrat variieren, aber sie liegen alle in einem engen Bereich (z. B. Arg131 DpIDH und Arg133 PcIDH, Tyr138 DpIDH und Tyr140 PcIDH).
- Das Metallion (Mg2+, Mn2+) bindet sich über Wasserstoffbrücken an drei konservierte Aminosäuren. Zu diesen Aminosäuren gehören drei Aspartat-Reste.
- NAD+ und NADP+ binden innerhalb des aktiven Zentrums in vier Regionen mit ähnlichen Eigenschaften bei den IDH-Enzymen. Diese Regionen variieren, liegen aber in der Nähe von , , , und . Auch hier variieren die Regionen, aber die Nähe der Regionen ist konserviert.