Einführung
Züchter und Forscher sind gleichermaßen an Hybridisierung und Introgression zwischen divergenten Genotypen als potenzieller Ursache für die ökologische Divergenz der Nachkommenschaft interessiert (Rieseberg et al., 2007; Arnold et al., 2012). Umgekehrt kann die Hybridisierung manchmal mit einer verringerten Hybridfitness einhergehen, wie z. B. Schwäche und/oder Sterilität in der F1-Generation und in späteren Generationen, selbst in Fällen von Hybridisierung zwischen Mitgliedern derselben Art.
Eine verringerte Hybridlebensfähigkeit und/oder Fruchtbarkeit, die in der F2-Generation oder in späteren Generationen segregiert, wird als Hybrid Breakdown (HB) bezeichnet, wobei rezessive Allele zwangsläufig damit verbunden sind. Diese Fortpflanzungsbarriere wird seit langem sowohl bei Pflanzen als auch bei Tieren beobachtet (Dobzhansky, 1970; Grant, 1971). Viele der genetischen Analysen dieser Barriere haben ergeben, dass sie durch einen komplementären Effekt zwischen und/oder unter Loci mit differenzierten Allelen zustande kommt, der gemeinhin als Bateson-Dobzhansky-Muller (BDM)-Inkompatibilität bezeichnet wird (Rieseberg und Willis, 2007). HB beinhaltet notwendigerweise intrinsische postzygotische Reproduktionsbarrieren, wie z. B. Hybridinvalidität (einschließlich Schwäche, Nekrose und Chlorose) und Hybridsterilität (in den männlichen, weiblichen oder beiden Gameten); daher unterscheiden manche Forscher HB nicht von Invalidität und Sterilität in der F1-Nachkommenschaft. Dennoch bin ich der Meinung, dass HB eine geeignete Klassifizierung für Reproduktionsbarrieren ist, da sie die zugrunde liegende genetische Basis impliziert (d. h. die Beteiligung rezessiver Allele).
Neuere Studien mit der Modellpflanze Arabidopsis haben zu einem besseren Verständnis von HB hinsichtlich ihrer molekularen Mechanismen geführt (Vaid und Laitinen, 2019). Obwohl Reis (Oryza-Arten) eine Modellpflanze ist, ist das derzeitige Verständnis der genetischen Grundlagen von HB bei dieser Art nach wie vor begrenzt. Dies liegt wahrscheinlich daran, dass HB bei F1-Hybriden, bei denen im Vergleich zu den elterlichen Inzuchtlinien eine höhere Kornproduktion erwartet wird, kein Fortpflanzungshindernis darstellt, und wahrscheinlich daran, dass minderwertige Pflanzen, die sich in der F2-Generation und in späteren Generationen segregieren, auf der Grundlage des Phänotyps aus der Zuchtpopulation leicht ausgewählt werden können. Die Schwierigkeit der genetischen Kartierung, die durch rezessive Vererbung verursacht wird, kann auch der Grund für diese Einschränkung sein.
Hier skizziere ich HB in Reiskreuzungen unter Bezugnahme auf Informationen, die von Arabidopsis-Studien geliefert wurden, und diskutiere, wie HB in der Reiszüchtung behandelt wird.
The Genetic Basis of Hybrid Breakdown in Rice
Es wird davon ausgegangen, dass kultivierter Reis, O. sativa japonica und indica, einen Artenkomplex mit ihrem mutmaßlichen Vorläufer (O. rufipogon) und wilden Arten bildet; jedoch werden bei Kreuzungen zwischen ihnen mehrere Reproduktionsbarrieren, einschließlich HB, beobachtet (Oka, 1988; Vaughan et al, 2003).
Meines Wissens war Dr. Oka der erste, der die genetische Grundlage der HB bei intersubspezifischen Kreuzungen zwischen Japonica und Indica beschrieb, die durch zwei komplementäre Gene erklärt wurde (Oka, 1957). Die verantwortlichen Gene wurden jedoch nicht auf Chromosomen kartiert, da zu dieser Zeit noch keine molekularen Marker verfügbar waren.
In Reishybriden wurde HB häufig bei intersubspezifischen (O. sativa ssp. japonica × ssp. indica) und interspezifischen (O. sativa × O. nivara und O. sativa × O. glumaepatula) Kreuzungen beschrieben (Oka, 1957; Sato und Morishima, 1988; Wu et al., 1995; Li et al., 1997; Fukuoka et al., 1998, 2005; Kubo und Yoshimura, 2002, 2005; Matsubara et al., 2007a,b, 2015; Yamamoto et al., 2007; Ichitani et al., 2012; für intersubspezifische Kreuzungen; Sobrizal et al., 2001; Miura et al., 2008 für interspezifische Kreuzungen) (Tabelle 1). Die meisten dieser HB-Fälle wurden durch Zwei-Lokus-BDM-Inkompatibilität und rezessive Allele verursacht, wobei 1/16 der F2-Nachkommenschaft, die an beiden Loci homozygot für rezessive Allele war, den HB-Phänotyp zeigte; jedoch zeigten 4/16 der Nachkommenschaft, die nur an einem Locus heterozygot war, den HB-Phänotyp, abhängig von der Kreuzungskombination (Tabelle 1 und Abbildung 1A). Bei den Rückkreuzungshybriden war ein Locus bereits mit den Allelen des wiederkehrenden Elternteils fixiert; daher wies 1/4 der BCnF2-Nachkommenschaft den HB-Phänotyp auf (Tabelle 1 und Abbildung 1B).

Table 1. Genetische Grundlage des Hybridabbaus bei Reiskreuzungen.
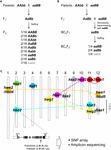
Abbildung 1. Die genetische Grundlage des Hybridabbaus (HB). (A) Schematische Darstellung des HB-Genotyps, der in der F2-Nachkommenschaft segregiert. Wenn die elterlichen Genotypen AAbb und aaBB sind, zeigt 1/16 der F2-Nachkommenschaft ohne dominantes Allel, das durch graue Zeichen angezeigt wird (d.h. aabb), eine reduzierte Lebensfähigkeit und/oder Fruchtbarkeit, obwohl die anderen Genotypen normal sind. Bei einigen Reiskreuzungen zeigen die F2-Nachkommen mit nur einem dominanten Allel (d. h. Aabb, aaBb) ebenfalls eine verringerte Lebensfähigkeit und/oder Fruchtbarkeit. (B) Schematische Darstellung des HB-Genotyps, der in der BCnF2-Nachkommenschaft segregiert. Außerdem zeigt 1/4 der BCnF2-Nachkommenschaft eine verminderte Lebensfähigkeit und/oder Fruchtbarkeit. (C) Chromosomale Lage der Gene, die HB in Reiskreuzungen zugrunde liegen. Die Gene sind auf der Grundlage der Ergebnisse der Genkartierung in den einzelnen Studien grob kartiert. Eine Reihe von komplementären Genen ist durch die gepunktete Linie verbunden. Komplementäre Gene mit hbd1 und hwf1 wurden nicht berichtet. Das Beispiel des funktionellen Nukleotid-Polymorphismus, der HB verursacht, wurde von Yamamoto et al. (2010) veröffentlicht, und diese Informationen werden es uns ermöglichen, eine Untersuchung mittels SNP-Array oder Amplikon-Sequenzierung durchzuführen.
Der HB-Phänotyp in Reishybriden variiert je nach Kreuzungskombination. Zum Beispiel zeigt der Genotyp hbd2/hbd2 hbd3/hbd3 Schwäche, aber keine offensichtliche Samensterilität. Der Genotyp hbd4/hbd4 hbd5/hbd5 hingegen weist sowohl Schwäche als auch Samensterilität auf (Matsubara et al., 2007a, 2015; Yamamoto et al., 2007; Tabelle 1). Es gibt auch komplizierte Fälle, in denen HB, die sowohl Schwäche als auch Samensterilität zeigen (Genotyp hwe1/hwe1 hwe2/hwe2), oder HB, die nur Samensterilität zeigen (hsa1/hsa1 hsa2/hsa2 hsa3/hsa3), trotz der gleichen Kreuzungskombination, d. h. Asominori × IR24-Kreuzung (Kubo und Yoshimura, 2002, 2005; Tabelle 1), beobachtet werden. Da diese HBs unabhängige genetische Grundlagen haben, würden mindestens 5/64 der F2-Nachkommenschaft den HB-Phänotyp in dieser Kreuzung zeigen.
Bis heute wurden mehrere Loci, die für HB verantwortlich sind, mit Hilfe von DNA-Markern bei Kreuzungen zwischen Japonica- und Indica-Reissorten in bestimmten genomischen Regionen kartiert (Chrs 7 und 10, Fukuoka et al, 1998; Chrs 1 und 12, Wu et al., 1995; Kubo und Yoshimura, 2002; Matsubara et al., 2015; Chrs 6 und 11, Fukuoka et al., 2005; Chrs 8, 9 und 12, Kubo und Yoshimura, 2005; Chrs 2 und 11, Matsubara et al., 2007a; Yamamoto et al., 2007, 2010; Chrs 11 und 12, Ichitani et al., 2012; Tabelle 1 und Abbildung 1C). Die Ergebnisse dieser Studien zeigten, dass die Loci, die der Reis-HB zugrunde liegen, in einigen Kreuzungen gemeinsam vorkommen, sich aber in anderen Kreuzungen unterscheiden, was auf ihre Diversifizierung in Reisgenomen hindeutet. Zum Beispiel teilt in einigen Fällen das HB-Allel von O. nivara, einer eng mit den Incica-Sorten verwandten Art, denselben Locus mit einer Indica-Sorte; das HB-Allel von O. glumaepatula, einer eng mit den Kultursorten verwandten Art, teilt jedoch den Locus mit keiner anderen Sorte (Tabelle 1 und Abbildung 1C). Es ist anzumerken, dass die oben beschriebenen HB-Fälle durch eine Reihe von Genen mit größeren Auswirkungen verursacht werden; es gibt jedoch auch Fälle, die durch eine Reihe von Genen mit geringeren Auswirkungen verursacht werden, z. B. eine leicht verringerte Samenfruchtbarkeit, die in der Reiszüchtung nicht übersehen werden können, die aber noch nicht entdeckt wurden.
Molecular Mechanisms Underlying Hybrid Breakdown
Autoimmunreaktion: Bomblies et al. (2007) berichteten erstmals, dass die Autoimmunreaktion, die ein NB-LRR-Krankheitsresistenzgen oder ein R-Gen auslöst, mit der HB von Arabidopsis in intraspezifischen Kreuzungen verbunden ist, obwohl diese Autoimmunreaktion hauptsächlich als Nekrose in F1-Pflanzen zum Ausdruck kommt und die HB in F2-Nachkommenschaft durch die Temperatur bedingt zu sein scheint. Bei Arabidopsis-Hybriden wurden mehrere Anzeichen für eine durch die Autoimmunreaktion verursachte HB beschrieben (Alcázar et al., 2009, 2010). Die Beteiligung von NB-LRR-Genen an der HB deutet darauf hin, dass mehrere genomische Regionen mit der Arabidopsis-HB in Verbindung gebracht werden können; in der Tat unterstützt die von Chae et al. (2014) durchgeführte umfassende Untersuchung diese Idee.
Auch bei Reishybriden wurde eine durch eine Autoimmunreaktion verursachte HB in einer Japonica × Indica-Kreuzung berichtet (Yamamoto et al., 2010). In diesem Fall tritt HB in F2-Pflanzen auf, wenn das Gen Hybrid Breakdown 2 (hbd2), das für Kaseinkinase I kodiert und von der Indica-Sorte getragen wird, mit einem Cluster von R-Genen kombiniert wird, die von der Japonica-Sorte getragen werden. Da sich diese verursachenden Allele wahrscheinlich teilweise rezessiv verhalten, können die Genprodukte daran beteiligt sein (Matsubara et al., 2007a; Yamamoto et al., 2007, 2010).
Reziprokes Silencing von duplizierten Genen: Dieser Gedanke wurde erstmals theoretisch als genetische Grundlage für die Hybridinkompatibilität vorgeschlagen (Werth und Windham, 1991; Lynch und Force, 2000). In diesem Modell verliert eine Population die Funktion an einem Locus und behält sie an dem anderen, während die andere Population den gegenteiligen Effekt erfährt. Folglich haben 1/16 der F2-Zygoten keine funktionalen Gene (Lynch und Force, 2000). Die von Bikard et al. (2009) und Vlad et al. (2010) beschriebene HB in Arabidopsis-Hybriden ist ein gutes Beispiel für dieses Phänomen. In diesem Szenario wurden auch Fälle von epigenetischem Silencing berichtet (Agorio et al., 2017; Blevins et al., 2017).
Da bei Reis sowohl eine Ganzgenom- als auch eine segmentale Duplikation stattgefunden hat (Wang et al., 2007; Guo et al., 2019), könnte reziprokes Silencing eine häufige Ursache für Hybridinkompatibilität sein. Bislang gibt es jedoch keine Hinweise auf HB, die durch reziprokes Silencing von duplizierten Genen in Reis verursacht wird.
Unabhängig von der Autoimmunreaktion und dem reziproken Silencing wurde eine Interaktion zwischen einer DEAD-Box-RNA-Helikase 18 und einem anderen Gen (MORPHEUS MOLECULE 1 als wahrscheinlicher Kandidat) identifiziert, die HB in Arabidopsis-Hybriden verursacht (Plötner et al., 2017; Vaid et al., 2020). Interessanterweise nimmt der HB-Phänotyp zwischen den F3- und F4-Generationen ab, was auf eine epigenetische Regulierung der Genexpression hindeutet.
In Reis-Hybriden haben Kubo et al. (2016) kürzlich gezeigt, dass der hsa1-Locus aus zwei Genen besteht und dass diese Gene für ein DUF1618-Protein bzw. ein nicht charakterisiertes Protein mit einer gewissen Ähnlichkeit zu einem Nukleotid-bindenden Protein kodieren. Die molekularen Eigenschaften der verbleibenden komplementären Gene, hsa2 und hsa3, sind nicht bekannt.
Hybridzusammenbruch in der konventionellen Reiszüchtung
Jahrzehntelang wurde die Bulk-Population-Methode in der konventionellen Reiszüchtung eingesetzt. Bei dieser Methode wird nach der Kreuzung die F4- oder F5-Population durch Selbstbefruchtung in der Masse (anstelle der Single-Seed-Descent-Methode) ohne künstliche Selektion gezüchtet, aber die Population der ersten Generation wird einer natürlichen Selektion und einer Selektion auf Lebensfähigkeit unterzogen (Allard, 1960; Ikehashi und Fujimaki, 1980 für Einzelheiten). Bei der Bulk-Population-Methode ist zu erwarten, dass viele schwache und/oder sterile Genotypen aus der Population eliminiert werden, bevor eine Population der fortgeschrittenen Generation entsteht; daher kann diese Methode nur begrenzte Informationen über HB liefern.
Zur Introgression oder Akkumulation wünschenswerter Merkmale von Spender(n) in eine Sorte haben Reiszüchter häufig Rückkreuzungen oder Kreuzungen mit mehreren Elternteilen durchgeführt, gefolgt von der Bulk-Population-Methode. Empirisch ist seit langem bekannt, dass diese Kreuzungsmethoden den Verlust von Selektionskandidaten durch Reproduktionsbarrieren abmildern können, da diese Methoden oft die Segregation von störenden Allelkombinationen, die mit HB in einer Hybridnachkommenschaft verbunden sind, reduzieren. Die Abschwächung von Reproduktionsbarrieren durch diese Kreuzungsmethoden hängt jedoch zwangsläufig vom HB-Genotyp der Eltern ab.
Hybridzusammenbruch in der genomikgestützten Züchtung
Die Veröffentlichung von Referenzpflanzengenomsequenzen und die Entwicklung von Sequenzierungstechnologien der nächsten Generation haben den Fortschritt der molekularen Züchtung von Nutzpflanzen beschleunigt (Kole et al., 2015). In diesem Zusammenhang wurde die genomikgestützte Züchtung, wie die genomische Selektion auf der Grundlage von Genotypen genomweiter DNA-Marker, in der Pflanzenzüchtung in Betracht gezogen (Spindel und Iwata, 2018).
In der genomikgestützten Züchtung selbstbefruchtender Nutzpflanzen werden häufig Populationen der fortgeschrittenen Generation, wie rekombinante Inzuchtlinien, als Referenzpopulationen verwendet, aus denen genomweite Genotyp- und Phänotypdaten gewonnen werden. Anschließend wird in den Nachkommen der frühen Generationen (z. B. F2) eine Selektion nur auf der Grundlage von Markergenotypen durchgeführt. Bei diesem Selektionsschema ist zu beachten, dass die Referenzpopulation in der Regel keine Informationen über die genomische Region für HB liefert, da schwache und/oder sterile Nachkommen in den ersten Generationen nach der Kreuzung eliminiert worden sein sollten. Wenn ein solches Selektionsschema angewandt wird, müssen wir daher einige wichtige Selektionskandidaten aufgeben. Alternativ könnten wir unerwünschte Kandidaten auswählen, so dass HB in späteren Generationen sichtbar wird.
Diskussion
Trotz der begrenzten Anzahl von Studien zu diesem Thema scheint es, dass die Loci, die HB in Reiskreuzungen zugrunde liegen, eher diversifiziert als geteilt sind (Tabelle 1 und Abbildung 1). Wie oben beschrieben, sollte die Information über die Verteilung der HB-assoziierten Allele unter den Kreuzungseltern eine Voraussetzung für die Reiszüchtung sein, insbesondere für die Maximierung der Effektivität der genomunterstützten Züchtung. Daher sind der zusätzliche Nachweis von HB in Reiskreuzungen und die Kartierung der verantwortlichen Gene im Reisgenom erforderlich. Bei Arabidopsis-Hybriden wurden solche Daten zu den R-Genen und ihren interagierenden Genen umfassend untersucht (Alcázar et al., 2010, 2014; Chae et al., 2014). Der Bericht über das hbd2-Gen von Yamamoto et al. (2010) in einer Reishybride ist ein gutes Beispiel für eine umfassende Erhebung, die mit SNP-Arrays oder Amplikonsequenzierung durchgeführt wurde (Abbildung 1C). Die Gene hbd3 und hsa1 sind ebenfalls Kandidaten für diese Art von Untersuchung. Letztendlich wird die Entwicklung von Kriterien, die es uns ermöglichen, HB auf der Grundlage der genomischen Informationen der Elternlinien vorherzusagen, ein wichtiges Instrument für die genomisch unterstützte Züchtung sein. Selbst wenn ein ursächlicher Faktor nicht als einzelnes Gen identifiziert wurde, werden eng verknüpfte Marker, wie z. B. Einzelnukleotid-Polymorphismen, für die Klassifizierung von HB-assoziierten Haplotypen wirksam sein, da das Kopplungsungleichgewicht von kultiviertem Reis schätzungsweise 100-200 kb beträgt, während es bei Wildarten (wie O. rufipogon) kann sich über mehrere Dutzend kb erstrecken (Mather et al., 2007; McNally et al., 2009; Huang et al., 2010). Diese Informationen werden es ermöglichen, eine effizientere und effektivere Kreuzungskombination zu entwerfen.
Die Analyse von Segreganten, gefolgt von der Sequenzierung der nächsten Generation, kann für die Kartierung von HB-assoziierten Loci sowie für die konventionelle Kopplungskartierung (z. B. quantitative Trait-Locus-Analyse) nützlich sein, da HB-Segreganten oft unterscheidbare Merkmale (über Merkmale wie Pflanzenhöhe, Anzahl der Pollen und Fruchtbarkeit) von normal wachsenden Segreganten in jeder Kreuzungspopulation aufweisen. Die Erfassung von Bilddaten mit einer Digitalkamera und einer Drohne kann ebenfalls eine wichtige Rolle bei der Phänotypisierung im Labor und auf dem Reisfeld spielen. Diese Bemühungen werden nicht nur Reiszüchtern, sondern auch Evolutionsbiologen wertvolle Informationen liefern.
Wird darüber hinaus ein HB-assoziiertes Allel identifiziert, kann eine Genunterbrechung durch Ionenstrahlen oder Genom-Editierung helfen, HB zu überwinden, wie das Beispiel der F1-Hybridsterilität bei Reis zeigt (Koide et al., 2018; Xie et al, 2019).
Datenverfügbarkeitserklärung
Alle in dieser Studie präsentierten Datensätze sind im Artikel/Ergänzungsmaterial enthalten.
Autorenbeiträge
Der Autor bestätigt, dass er der einzige Autor dieser Arbeit ist und sie zur Veröffentlichung freigegeben hat.
Interessenkonflikt
Die Autoren erklären, dass die Forschung in Abwesenheit jeglicher kommerzieller oder finanzieller Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagungen
Ich möchte diese Arbeit dem Andenken von Dr. Darshan Brar widmen. Er war ein hervorragender Reiszüchter, der energisch nützliche Gene, die von Wildarten getragen wurden, in Kultursorten jenseits von Reproduktionsbarrieren einführte.
Allard, R. W. (1960). Principles of Plant Breeding. New York, NY: John Wily and Sons, Inc.
Google Scholar
Dobzhansky, T. (1970). Genetics of the Evolutionary Process. New York, NY: Columbia University Press.
Google Scholar
Grant, V. (1971). Plant Speciation. New York, NY: Columbia University Press.
Google Scholar
Ikehashi, H., and Fujimaki, H. (1980). „Modified bulk population method for rice breeding,“ in Proceedings of the International Rice Research Conference on Innovative approaches to rice breeding, (Los Baños, PA: International Rice Research Institute), 163-182.
Google Scholar
Oka, H.-I. (1988). Origin of Cultivated Rice. Tokyo: JSSP.
Google Scholar