Glykogen und Stärke, Glukosepolymere von Megadalton-Größe, sind in den meisten lebenden Organismen das Hauptreservoir an schnell verfügbarer Energie und Kohlenstoffverbindungen. Bei Säugetieren unterliegen die Aufnahme und Verwertung von Glukose einer strengen Kontrolle. Defekte in der normalen Glukoseverarbeitung werden mit einer Reihe menschlicher Krankheiten in Verbindung gebracht, wie z. B. Glykogenspeicherkrankheiten und Diabetes, bei denen eine anhaltende Hyperglykämie mit einem frühen Ausbruch und einem höheren Schweregrad der Krankheit einhergeht. An den Stoffwechselwegen für die katalytische Umwandlung von Glykogen sind mehrere Enzyme beteiligt, von denen die Glykogensynthase den Aufbau und die Glykogenphosphorylase den Abbau von a(1,4)-verknüpften Glucanen katalysiert. In den letzten 30 Jahren wurde durch biochemische und strukturelle Untersuchungen von Glykogen- und Maltodextrinphosphorylasen eine Fülle von Informationen über den Glykogenabbau gewonnen. Diese Studien haben gezeigt, dass die Regulierung der Glykogenphosphorylase ein faszinierendes Beispiel für kovalenten und nicht-kovalenten Allosterismus ist, der durch hormonelle Kontrolle hervorragend koordiniert wird. Für praktisch alle Enzyme, die direkt an der Glykogenverarbeitung beteiligt sind, liegen jetzt Informationen über die tertiären und quaternären Strukturen vor. Überraschenderweise gibt es nur sehr wenige Strukturinformationen über die Glykogensynthase, die die Glykogenverlängerung durch die sukzessive Addition von ![]() -(1,4)-verknüpften Glukoseresten an das nichtreduzierende Ende des Polymers katalysiert, wobei ADP/UDP-Glukose als Glukosedonor verwendet wird.
-(1,4)-verknüpften Glukoseresten an das nichtreduzierende Ende des Polymers katalysiert, wobei ADP/UDP-Glukose als Glukosedonor verwendet wird.
Wir haben jetzt die Kristallstruktur der Glykogensynthase von Agrobacterium tumefaciens (AtGS) mit einer Auflösung von 2,3 Å bestimmt, sowohl in ihrer apo-Form als auch in Gegenwart von ADP. Die Daten wurden mit ID29 gesammelt und die Struktur mit der SAD-Technik (anomale Einzelwellenlängenbeugung) und anschließenden Dichtemodifikationsverfahren gelöst. Das Protein liegt als Dimer in der asymmetrischen Einheit vor, wobei jedes Monomer aus zwei „Rossmann-fold“-Domänen besteht (Abbildung 81), die wie in den Strukturen der Glykogenphosphorylase und anderer Glykosyltransferasen der GT-B-Superfamilie angeordnet sind. Ein tiefer Spalt zwischen den beiden Domänen enthält das katalytische Zentrum. Ein wesentlicher Unterschied zwischen Glykogensynthasen von Säugetieren (Familie GT3) und Bakterien (GT5) besteht darin, dass nur GT3-Enzyme durch Phosphorylierung und nicht-kovalente allosterische Modulation streng reguliert werden. Nach den strukturbasierten multiplen Sequenzalignments zwischen GT3-, GT5- und Glykogenphosphorylase-Sequenzen befinden sich die regulatorischen Phosphorylierungsstellen in der menschlichen Glykogensynthase wahrscheinlich in den N-terminalen Insertionen mit 25 Residuen und den C-terminalen Insertionen mit 120 Residuen, außerhalb des nicht regulierten GT-B-Kerns der bakteriellen Glykogensynthasen. Mutagenese-Studien hatten zuvor zwei konservierte Arg-Cluster in GT3-Enzymen als Teil der allosterischen Stelle(n) sowohl für die Phospho-Ser/Thr-Reste als auch für den nicht-kovalenten Aktivator Glc-6P identifiziert. Die entsprechenden Reste in AtGS befinden sich an einem Ende der C-terminalen Domäne, weit entfernt vom Reaktionszentrum, was auf eine weitreichende regulatorische Wirkung hinweist.
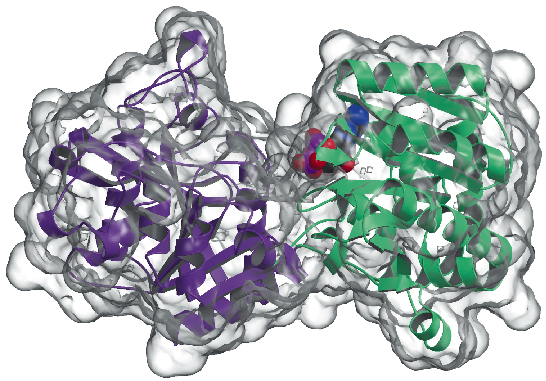
Abbildung 81: Struktur der Glykogensynthase: Die lösungsmittelzugängliche Oberfläche ist transparent dargestellt. Man beachte das gebundene ADP-Molekül (als VDW-Kugeln gezeichnet).
Die Struktur von AtGS zeigt eine auffallende topologische Ähnlichkeit mit den Kernstrukturen der Glykogen/Maltodextrin-Phosphorylasen (Familie GT35) (Abbildung 82a), obwohl die Sequenzidentitäten gering sind (~14%). Die Struktur jeder Rossmann-Faltdomäne von AtGS liegt deutlich näher an der Struktur von Phosphorylasen der GT35-Familie als an der Struktur jeder anderen nukleotidabhängigen Glykosyltransferase. Glykogensynthase und Phosphorylasen weisen auch eine starke Ähnlichkeit an ihren katalytischen Stellen auf (Abbildung 82b). Die ADP-Ribose und die Pyridoxalgruppen befinden sich an den gleichen Stellen, ebenso wie das distale Phosphat von ADP in AtGS und das anorganische Phosphatsubstrat in Phosphorylasen. Darüber hinaus sind die kritischen Aminosäuren, die mit den Glukoseeinheiten der Substrate interagieren, ebenfalls konserviert. Der katalytische Mechanismus von Glykogensynthasen und Phosphorylasen ist daher wahrscheinlich ähnlich, wobei subtile Änderungen in der Struktur ihre insgesamt unterschiedlichen funktionellen Eigenschaften erklären.
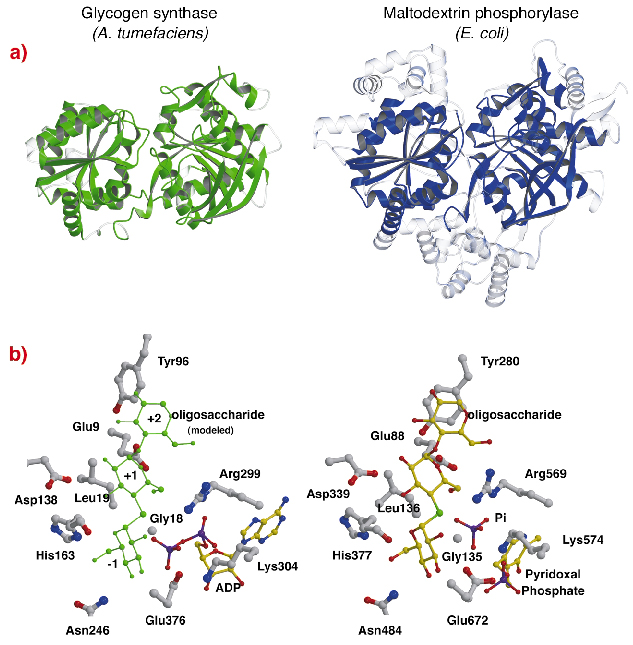
Abb. 82: Homologie zur Glykogenphosphorylase: (a) nach strukturellem Alignment, überlagerte äquivalente Reste zwischen AtGS und E. coli Maltodextrinphosphorylase MalP, dargestellt in festem Material (nicht ausgerichtete Regionen, in transparentem); (b) die katalytischen Stellen von AtGS und MalP.
Die ursprüngliche Annahme, dass die Glykogensynthese einfach eine Umkehrung der abbauenden Phosphorolyse darstellt, erwies sich als falsch, als Leloir zeigte, dass UDP-Glc und die Glykogensynthase, nicht Glc-1-P und die Glykogenphosphorylase, für die Polysaccharidverlängerung verantwortlich sind. Unsere Arbeit zeigt nun jedoch, dass die entgegengesetzten unabhängigen Reaktionen bei der Glukose-Glykogen-Umwandlung durch eng verwandte homologe Enzyme katalysiert werden.