El glucógeno y el almidón, polímeros de glucosa del tamaño de un megadalton, son la principal reserva de energía y compuestos de carbono fácilmente disponibles en la mayoría de los organismos vivos. En los mamíferos, la captación y la utilización de la glucosa están muy controladas. Los defectos en el manejo normal de la glucosa se asocian a diversas patologías humanas, como las enfermedades de almacenamiento de glucógeno y la diabetes, en las que la hiperglucemia persistente se correlaciona con una aparición temprana y una mayor gravedad de la enfermedad. Las vías metabólicas para la conversión catalítica del glucógeno implican varias enzimas, entre las que la glucógeno sintasa cataliza la elongación, y la glucógeno fosforilasa la descomposición, de los glucanos ligados a(1,4). En los últimos 30 años se ha obtenido una gran cantidad de información sobre la degradación del glucógeno a través de los estudios bioquímicos y estructurales de las fosforilasas del glucógeno y la maltodextrina. Estos estudios han demostrado que la regulación de la glucógeno fosforilasa es un ejemplo fascinante de alosterismo covalente y no covalente, exquisitamente coordinado por el control hormonal. Actualmente se dispone de información que describe las estructuras terciarias y cuaternarias de prácticamente todas las enzimas que participan directamente en el procesamiento del glucógeno. Sorprendentemente, se ha comunicado muy poca información estructural sobre la glucógeno sintasa, que cataliza la elongación del glucógeno mediante la adición sucesiva de residuos de glucosa ![]() -(1,4)-vinculados al extremo no reductor del polímero, utilizando ADP/UDP-glucosa como donante de glucosa.
-(1,4)-vinculados al extremo no reductor del polímero, utilizando ADP/UDP-glucosa como donante de glucosa.
Ahora hemos determinado la estructura cristalina de la glucógeno sintasa de Agrobacterium tumefaciens (AtGS) a una resolución de 2,3 Å, tanto en su forma apo como en presencia de ADP. Los datos se recogieron en ID29, y la estructura se resolvió mediante la técnica SAD (difracción anómala de longitud de onda única), seguida de procedimientos de modificación de la densidad. La proteína se encuentra como un dímero en la unidad asimétrica, con cada monómero compuesto por dos dominios «Rossmann-fold» (Figura 81) que se organizan como en las estructuras de la glucógeno fosforilasa y otras glucosil transferasas de la superfamilia GT-B . Una profunda fisura entre ambos dominios contiene el centro catalítico. Una diferencia importante entre las glucógeno sintasas de mamíferos (familia GT3) y las bacterianas (GT5) es que sólo las enzimas GT3 están estrechamente reguladas por fosforilación y modulación alostérica no covalente. Según las alineaciones de secuencias múltiples basadas en la estructura entre las secuencias de GT3, GT5 y la glicógeno fosforilasa, los sitios de fosforilación reguladores en la glicógeno sintasa humana están probablemente localizados en las inserciones N-terminal de 25 residuos y C-terminal de 120 residuos, fuera del núcleo GT-B no regulado de las glicógeno sintasas bacterianas. Los estudios de mutagénesis habían identificado previamente dos grupos de Arg conservados en las enzimas GT3 como parte del sitio(s) alostérico(s) tanto para los residuos fosfo-Ser/Thr como para el activador no covalente Glc-6P. Los residuos equivalentes en AtGS están situados en un extremo del dominio C-terminal, lejos del centro de reacción, lo que indica un efecto regulador a larga distancia.
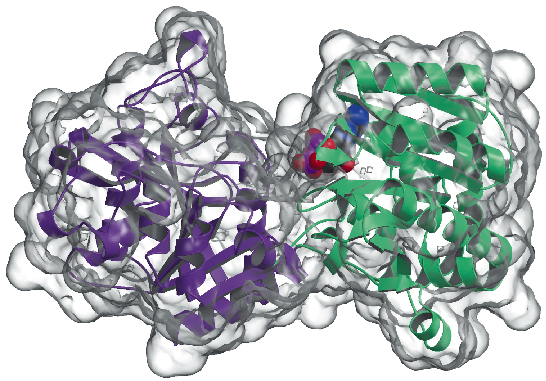
Fig. 81: Estructura de la glucógeno sintasa: la superficie accesible al disolvente se representa en transparente. Obsérvese la molécula de ADP unida (dibujada como bolas VDW).
La estructura de la AtGS muestra un sorprendente parecido topológico con las estructuras del núcleo de las fosforilasas de glucógeno/maltodextrina (familia GT35) (Figura 82a), aunque las identidades de secuencia son bajas (~14%). La estructura de cada dominio del pliegue de Rossmann de AtGS es significativamente más cercana a la de las fosforilasas de la familia GT35 que a la de cualquier otra glucosil transferasa dependiente de nucleótidos. La glucógeno sintasa y las fosforilasas también muestran un gran parecido en sus sitios catalíticos (Figura 82b). La ribosa ADP y los grupos piridoxal se encuentran en posiciones equivalentes, al igual que el fosfato distal del ADP en la AtGS y el sustrato de fosfato inorgánico en las fosforilasas. Además, también se conservan los aminoácidos críticos que interactúan con los restos de glucosa de los sustratos. El mecanismo catalítico utilizado por las glucógeno sintasas y las fosforilasas es, por tanto, probablemente similar, con sutiles cambios en la estructura que explican sus diferentes propiedades funcionales generales.
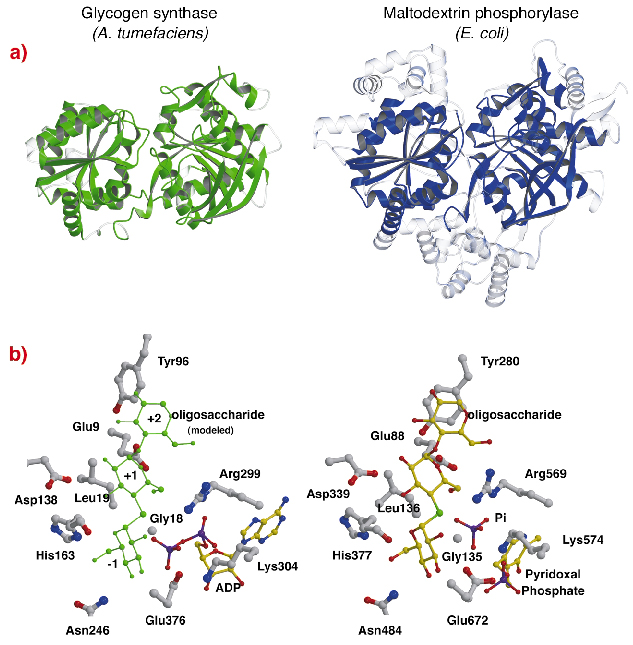
Fig. 82: Homología con la glucógeno fosforilasa: (a) tras el alineamiento estructural, los residuos equivalentes superpuestos entre AtGS y la maltodextrina fosforilasa MalP de E. coli, se representan en material sólido (las regiones no alineadas, en transparente); (b) los sitios catalíticos de AtGS y MalP.
La sugerencia inicial de que la síntesis de glucógeno representaba simplemente una inversión de su fosforólisis degradativa se demostró que era errónea, ya que Leloir demostró que el UDP-Glc y la glucógeno sintasa, y no el Glc-1-P y la glucógeno fosforilasa, eran responsables de la elongación del polisacárido . Nuestro trabajo demuestra ahora, sin embargo, que las reacciones independientes opuestas en la interconversión glucosa-glicógeno son catalizadas por enzimas homólogas estrechamente relacionadas.