- Introducción
- La base genética de la ruptura de híbridos en el arroz
- Mecanismos moleculares que subyacen a la ruptura del híbrido
- Destrucción de híbridos en el cultivo convencional del arroz
- La ruptura de híbridos en la mejora genómica-asistida
- Discusión
- Declaración de disponibilidad de datos
- Contribuciones del autor
- Conflicto de intereses
- Agradecimientos
Introducción
Tanto los criadores como los investigadores se han interesado por la hibridación y la introgresión entre genotipos divergentes como el motor potencial que conduce a la divergencia ecológica de la progenie (Rieseberg et al., 2007; Arnold et al., 2012). Por el contrario, la hibridación puede implicar a veces una reducción de la aptitud híbrida, como debilidad y/o esterilidad en la F1 y generaciones posteriores, incluso en casos de hibridación entre miembros de la misma especie.
La reducción de la viabilidad híbrida y/o de la fertilidad que segrega en la F2 o en generaciones posteriores se denomina ruptura híbrida (HB), en la que necesariamente se asocian alelos recesivos. Esta barrera reproductiva se ha observado durante mucho tiempo tanto en plantas como en animales (Dobzhansky, 1970; Grant, 1971). Muchos de los análisis genéticos de esta barrera han revelado que se lleva a cabo por un efecto complementario entre y/o entre loci con alelos diferenciados, comúnmente llamado incompatibilidad Bateson-Dobzhansky-Muller (BDM) (Rieseberg y Willis, 2007). La HB implica necesariamente barreras reproductivas postzigóticas intrínsecas, como la inviabilidad híbrida (que incluye debilidad, necrosis y clorosis) y la esterilidad híbrida (en el macho, la hembra o ambos gametos); por lo tanto, algunos investigadores pueden no distinguir la HB de la inviabilidad y la esterilidad en la progenie F1. Sin embargo, creo que la HB es una clasificación conveniente para las barreras reproductivas porque implica su base genética subyacente (es decir, la participación de alelos recesivos).
Estudios recientes utilizando la especie de planta modelo Arabidopsis han proporcionado una mejor comprensión de la HB en cuanto a sus mecanismos moleculares (Vaid y Laitinen, 2019). Por el contrario, aunque el arroz (especie Oryza) es un cultivo modelo, la comprensión actual de las bases genéticas de la HB sigue siendo limitada en esta especie, probablemente porque la HB no es una barrera reproductiva en los híbridos F1, en los que se espera una mayor producción de grano en comparación con las líneas endocriadas parentales, y probablemente porque las plantas inferiores que segregan en la F2 y en las generaciones posteriores pueden ser fácilmente seleccionadas en base al fenotipo de la población reproductora. La dificultad en el mapeo genético causada por la herencia recesiva también puede estar detrás de esta limitación.
Aquí, esbozo el HB en los cruces de arroz mientras me refiero a la información proporcionada por los estudios de Arabidopsis, y discuto cómo se maneja el HB en la cría de arroz.
La base genética de la ruptura de híbridos en el arroz
Se considera que el arroz cultivado, O. sativa japónica e índica, forma un complejo de especies con su putativo progenitor (O. rufipogon) y las especies silvestres; sin embargo, se observan múltiples barreras reproductivas, incluyendo HB, en los cruces entre ellos (Oka, 1988; Vaughan et al., 2003).
Hasta donde yo sé, el Dr. Oka fue el primero en describir la base genética de la HB en los cruces interespecíficos entre japonica e indica, que se explicaba por dos genes complementarios (Oka, 1957). Sin embargo, los genes responsables no fueron mapeados en los cromosomas, porque los marcadores moleculares no estaban disponibles en ese momento.
En los híbridos de arroz, el HB se ha descrito a menudo en cruces interespecíficos (O. sativa ssp. japonica × ssp. indica) e interespecíficos (O. sativa × O. nivara y O. sativa × O. glumaepatula) (Oka, 1957; Sato y Morishima, 1988; Wu et al., 1995; Li et al., 1997; Fukuoka et al., 1998, 2005; Kubo y Yoshimura, 2002, 2005; Matsubara et al., 2007a,b, 2015; Yamamoto et al., 2007; Ichitani et al., 2012; para los cruces interespecíficos; Sobrizal et al., 2001; Miura et al., 2008 para los cruces interespecíficos) (Tabla 1). La mayoría de estos casos de HB fueron causados por la incompatibilidad BDM de dos locus y alelos recesivos, por lo que 1/16 de la progenie F2 que era homocigota para alelos recesivos en ambos loci mostró el fenotipo HB; sin embargo, 4/16 de la progenie que era heterocigota en un solo locus mostró el fenotipo HB, dependiendo de la combinación de cruces (Tabla 1 y Figura 1A). En los híbridos retrocruzados, un locus ya estaba fijado con los alelos del progenitor recurrente; por lo tanto, 1/4 de la progenie BCnF2 mostró el fenotipo HB (Tabla 1 y Figura 1B).

Tabla 1. Base genética de la ruptura de híbridos reportada en los cruces de arroz.
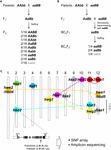
Figura 1. La base genética de la ruptura híbrida (HB). (A) Representación esquemática del genotipo HB que segrega en la progenie F2. Cuando los genotipos parentales son AAbb y aaBB, 1/16 de la progenie F2 sin alelo dominante, que se indica con el carácter gris (es decir, aabb), muestra una viabilidad y/o fertilidad reducida, aunque los otros genotipos son normales. En algunos cruces de arroz, la progenie F2 con un solo alelo dominante (es decir, Aabb, aaBb) también muestra una viabilidad y/o fertilidad reducidas. (B) Representación esquemática del genotipo HB que segrega en la progenie BCnF2. Además, 1/4 de la progenie BCnF2 muestra una viabilidad y/o fertilidad reducida. (C) Localización cromosómica de los genes que subyacen al HB en los cruces de arroz. Los genes se han trazado a grandes rasgos basándose en los resultados del mapeo de genes de cada estudio. Un conjunto de genes complementarios están conectados por la línea de puntos. No se ha informado de los genes complementarios con hbd1 y hwf1. Se muestra el ejemplo del polimorfismo funcional de nucleótidos causante de HB publicado por Yamamoto et al. (2010), y esta información nos permitirá realizar un estudio mediante array de SNP o secuenciación de amplicones.
El fenotipo HB en los híbridos de arroz varía según la combinación de cruces. Por ejemplo, el genotipo hbd2/hbd2 hbd3/hbd3 muestra debilidad, pero no esterilidad evidente en las semillas. Sin embargo, el genotipo hbd4/hbd4 hbd5/hbd5 muestra tanto debilidad como esterilidad de las semillas (Matsubara et al., 2007a, 2015; Yamamoto et al., 2007; Tabla 1). También hay casos complicados en los que se observan HB que muestran tanto debilidad como esterilidad de las semillas (genotipo hwe1/hwe1 hwe2/hwe2) o HB que sólo muestran esterilidad de las semillas (hsa1/hsa1 hsa2/hsa2 hsa3/hsa3), a pesar de la misma combinación de cruces, es decir, el cruce Asominori × IR24 (Kubo y Yoshimura, 2002, 2005; Tabla 1). Como estos HB tienen bases genéticas independientes, al menos 5/64 de la progenie F2 mostrarían el fenotipo HB en este cruce.
Hasta la fecha, se han mapeado varios conjuntos de loci responsables de HB en regiones genómicas particulares utilizando marcadores de ADN en cruces entre las variedades de arroz japónica e índica (Chrs 7 y 10, Fukuoka et al., 1998; Chrs 1 y 12, Wu et al., 1995; Kubo y Yoshimura, 2002; Matsubara et al., 2015; Chrs 6 y 11, Fukuoka et al., 2005; Chrs 8, 9 y 12, Kubo y Yoshimura, 2005; Chrs 2 y 11, Matsubara et al., 2007a; Yamamoto et al., 2007, 2010; Chrs 11 y 12, Ichitani et al., 2012; Tabla 1 y Figura 1C). Los resultados de estos estudios revelaron que los loci subyacentes al HB del arroz son compartidos en algunos cruces, pero difieren en otros, lo que sugiere su diversificación en los genomas del arroz. Por ejemplo, en algunos casos el alelo HB portado por O. nivara, una especie estrechamente relacionada con las variedades incica, comparte el mismo locus con una variedad indica; sin embargo, el alelo HB portado por O. glumaepatula, una especie estrechamente relacionada con las variedades cultivadas, no comparte el locus con ninguna otra variedad (Tabla 1 y Figura 1C). Cabe señalar que los casos de HB descritos anteriormente están causados por un conjunto de genes con efectos mayores; sin embargo, también hay casos causados por un conjunto de genes con efectos menores, por ejemplo, una ligera reducción de la fertilidad de las semillas, que no pueden pasarse por alto en la mejora del arroz, que aún no se han detectado.
Mecanismos moleculares que subyacen a la ruptura del híbrido
Respuesta autoinmune: Bomblies et al. (2007) informaron por primera vez de que la respuesta autoinmune que un gen de resistencia a la enfermedad NB-LRR o gen R está asociada a la HB de Arabidopsis en cruces intraespecíficos, aunque esta respuesta autoinmune se expresa principalmente como necrosis en las plantas F1, y la HB en la progenie F2 parecía estar condicionada por la temperatura. En híbridos de Arabidopsis, se han descrito varias líneas de evidencia de HB causada por la respuesta autoinmune (Alcázar et al., 2009, 2010). La implicación de los genes NB-LRR en la HB sugiere que múltiples regiones genómicas pueden estar asociadas a la HB de Arabidopsis; de hecho, el amplio estudio realizado por Chae et al. (2014) apoya esta idea.
También en híbridos de arroz se ha descrito la HB causada por una respuesta autoinmune en un cruce japonica × indica (Yamamoto et al., 2010). En este caso, la HB se produce en las plantas F2 cuando el gen de la descomposición híbrida 2 (hbd2), que codifica la caseína quinasa I y es portador de la variedad índica, se combina con un grupo de genes R portadores de la variedad japónica. Como es probable que estos alelos causantes se comporten como parcialmente recesivos, los productos del gen pueden estar implicados (Matsubara et al., 2007a; Yamamoto et al., 2007, 2010).
Silenciamiento recíproco de genes duplicados: esta noción se propuso por primera vez de forma teórica como base genética de la incompatibilidad híbrida (Werth y Windham, 1991; Lynch y Force, 2000). En el modelo, una población pierde la función en un locus y la conserva en el otro, mientras que la otra población experimenta el efecto contrario. En consecuencia, 1/16 de los cigotos F2 no tienen genes funcionales (Lynch y Force, 2000). El HB en híbridos de Arabidopsis descrito por Bikard et al. (2009) y Vlad et al. (2010) es un buen ejemplo de este fenómeno. En este escenario, también se han reportado casos que involucran el silenciamiento epigenético (Agorio et al., 2017; Blevins et al., 2017).
Como el arroz ha experimentado tanto la duplicación de todo el genoma como la de segmentos (Wang et al., 2007; Guo et al., 2019), el silenciamiento recíproco podría ser prevalente como causa de incompatibilidad híbrida. Sin embargo, hasta la fecha, no hay evidencia de HB causada por el silenciamiento recíproco de genes duplicados en el arroz.
Independientemente de la respuesta autoinmune y el silenciamiento recíproco, se ha identificado una interacción entre una ARN helicasa DEAD-box 18 y otro gen (MORPHEUS MOLECULE 1 como probable candidato) que causa HB en los híbridos de Arabidopsis (Plötner et al., 2017; Vaid et al., 2020). Curiosamente, el fenotipo HB se reduce entre las generaciones F3 y F4, lo que implica una regulación epigenética de la expresión génica.
En los híbridos de arroz, entre los tres loci de esterilidad híbrida-a (hsa), Kubo et al. (2016) mostraron recientemente que el locus hsa1 consta de dos genes, y que estos genes codifican una proteína DUF1618 y una proteína no caracterizada con cierta similitud a una proteína de unión a nucleótidos, respectivamente. Las características moleculares de los restantes genes complementarios, hsa2 y hsa3, no han sido comunicadas.
Destrucción de híbridos en el cultivo convencional del arroz
Durante décadas, el método de población masiva fue ampliamente empleado en el cultivo convencional del arroz. En este método, tras el cruce, la población F4 o F5 se cría por autofecundación a granel (en lugar de por el método de descenso de una sola semilla) sin selección artificial, pero la población de la primera generación se somete a una selección natural y de viabilidad (Allard, 1960; Ikehashi y Fujimaki, 1980 para más detalles). En el método de población masiva, cabe esperar que muchos genotipos débiles y/o estériles sean eliminados de la población antes de que se establezca una población de generación avanzada; por lo tanto, este método puede proporcionar información limitada sobre la HB.
Para la introgresión o la acumulación de rasgos deseables de donante(s) a una variedad, los obtentores de arroz han realizado a menudo retrocruzamientos o cruces parentales múltiples, seguidos del método de población masiva. Empíricamente, se sabe desde hace tiempo que estos métodos de cruce permiten mitigar la pérdida de candidatos a la selección por barreras reproductivas, ya que estos métodos suelen reducir la segregación de combinaciones disruptivas de alelos asociadas a HB en una progenie híbrida. Sin embargo, la mitigación de las barreras reproductivas mediante estos métodos de cruce depende inevitablemente del genotipo HB de los progenitores.
La ruptura de híbridos en la mejora genómica-asistida
La publicación de las secuencias del genoma de los cultivos de referencia y el desarrollo de las tecnologías de secuenciación de nueva generación han acelerado el progreso de la mejora molecular de los cultivos (Kole et al., 2015). En este contexto, la mejora asistida por la genómica, como la selección genómica basada en genotipos de marcadores de ADN de todo el genoma, se ha considerado en la mejora de cultivos (Spindel e Iwata, 2018).
En la mejora asistida por la genómica de cultivos autógamos, las poblaciones de generación avanzada, como las líneas endógamas recombinantes, se suelen utilizar como poblaciones de referencia, de las que se obtienen datos de genotipo y fenotipo de todo el genoma. Posteriormente, la selección basada únicamente en los genotipos de los marcadores se lleva a cabo en la progenie de las primeras generaciones (por ejemplo, F2). En este esquema de selección, hay que tener en cuenta que la población de referencia no suele proporcionar la información de la región genómica para HB, porque la progenie débil y/o estéril debería haber sido eliminada en las primeras generaciones después del cruce. Por lo tanto, si se adopta este esquema de selección, tendremos que abandonar algunos candidatos de selección importantes. Alternativamente, podemos seleccionar candidatos indeseables de manera que la HB se haga evidente en generaciones posteriores.
Discusión
A pesar del limitado número de estudios sobre este tema, parece que los loci que subyacen a la HB en los cruces de arroz están diversificados más que compartidos (Tabla 1 y Figura 1). Como se ha descrito anteriormente, la información sobre la distribución de los alelos asociados a HB entre los progenitores de los cruces debería ser un requisito previo para la mejora del arroz, especialmente para maximizar la eficacia de la mejora asistida por la genómica. Por lo tanto, la detección adicional de HB en los cruces de arroz y el mapeo de los genes responsables en el genoma del arroz son necesarios. En los híbridos de Arabidopsis, estos datos sobre los genes R y sus genes interactivos han sido ampliamente estudiados (Alcázar et al., 2010, 2014; Chae et al., 2014). El informe sobre el gen hbd2 realizado por Yamamoto et al. (2010) en un híbrido de arroz es un buen ejemplo de un amplio estudio realizado mediante arrays de SNP o secuenciación de amplicones (Figura 1C). Los genes hbd3 y hsa1 también son candidatos para este tipo de estudio. Con el tiempo, el desarrollo de criterios que nos permitan predecir el HB en base a la información genómica de las líneas parentales servirá como una herramienta importante para la cría asistida por la genómica. Aunque no se haya identificado un factor causal como un solo gen, los marcadores estrechamente vinculados, como los polimorfismos de un solo nucleótido, serán eficaces para clasificar los haplotipos asociados al HB, ya que se ha estimado que el desequilibrio de enlaces del arroz cultivado se extiende hasta 100-200 kb, aunque el de las especies silvestres (como O. rufipogon) pueden extenderse por varias decenas de kb (Mather et al., 2007; McNally et al., 2009; Huang et al., 2010). Esta información permitirá el diseño de una combinación de cruces más eficiente y eficaz.
El análisis de segregantes seguido de la secuenciación de próxima generación puede ser útil para el mapeo de los loci asociados a HB, así como el mapeo de vinculación convencional (como el análisis de locus de rasgos cuantitativos), porque los segregantes de HB a menudo muestran características distinguibles (sobre rasgos como la altura de la planta, el número de tallos y la fertilidad) de los segregantes de crecimiento normal en cada población de cruce. La adquisición de datos de imágenes mediante una cámara digital y la carga de un dron también puede desempeñar un papel importante en el fenotipado en el laboratorio y en el campo de arroz. Estos esfuerzos proporcionarán información valiosa no solo a los criadores de arroz, sino también a los biólogos evolutivos.
Además, si se identifica un alelo asociado a la HB, la interrupción del gen a través del haz de iones o la edición del genoma puede ayudar a superar la HB, como se ejemplifica en la esterilidad híbrida F1 en el arroz (Koide et al., 2018; Xie et al, 2019).
Declaración de disponibilidad de datos
Todos los conjuntos de datos presentados en este estudio se incluyen en el artículo/material suplementario.
Contribuciones del autor
El autor confirma ser el único contribuyente de este trabajo y lo ha aprobado para su publicación.
Conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
Agradecimientos
Me gustaría dedicar este trabajo a la memoria del Dr. Darshan Brar. Fue un eminente criador de arroz que introdujo vigorosamente genes útiles portados por especies silvestres en cultivares más allá de las barreras reproductivas.
Allard, R. W. (1960). Principles of Plant Breeding. Nueva York, NY: John Wily and Sons, Inc.
Google Scholar
Dobzhansky, T. (1970). Genética del proceso evolutivo. New York, NY: Columbia University Press.
Google Scholar
Grant, V. (1971). Plant Speciation. New York, NY: Columbia University Press.
Google Scholar
Ikehashi, H., y Fujimaki, H. (1980). «Modified bulk population method for rice breeding», en Proceedings of the International Rice Research Conference on Innovative approaches to rice breeding, (Los Baños, PA: International Rice Research Institute), 163-182.
Google Scholar
Oka, H.-I. (1988). Origen del arroz cultivado. Tokio: JSSP.
Google Scholar