Isocitrato desidrogenase catalisa as reacções químicas:
Isocitrato + NAD+ ⇌

2-oxoglutarate + CO2 + NADH + H+ Isocitrate + NADP+ ⇌ {\i1}displaystyle {\i1}rightleftharpoons

2-oxoglutarato + CO2 + NADPH + H+
A energia total livre para esta reacção é -8,4 kJ/mol.
PassosEditar
No ciclo do ácido cítrico, o isocitrato, produzido a partir da isomerização do citrato, sofre tanto a oxidação como a descarboxilação. Usando a enzima isocitrato desidrogenase (IDH), o isocitrato é mantido dentro de seu local ativo pela arginina, tirosina, asparagina, serina, treonina e aminoácidos do ácido aspártico circundantes. A primeira caixa mostra a reação global da isocitrato desidrogenase. Os reagentes necessários para este mecanismo enzimático funcionar são isocitrato, NAD+/NADP+, e Mn2+ ou Mg2+. Os produtos da reação são o alfa-ketoglutarato, dióxido de carbono e NADH + H+/NADPH + H+. Moléculas de água são usadas para ajudar a desprotonificar os oxiógenos (O3) do isocitrato.
A segunda caixa é o Passo 1, que é a oxidação do alfa C (C#2). A oxidação é o primeiro passo pelo qual passa o isocitrato. Neste processo, o grupo álcool do alfa-carbono (C#2) é desprotonado e os electrões fluem para o alfa-C formando um grupo cetona e removendo um hidreto do C#2 usando NAD+/NADP+ como um cofactor de aceitação de electrões. A oxidação do alfa-C permite uma posição onde os elétrons (no próximo passo) estarão descendo do grupo carboxil e empurrando os elétrons (fazendo com que o oxigênio duplamente ligado) de volta para cima no oxigênio ou pegando um próton próximo de um aminoácido lisina.
A terceira caixa é o passo 2, que é a descarboxilação do oxalosuccinato. Nesta etapa, o oxigênio do grupo carboxil é desprotonado por um aminoácido de Tirosina próximo e esses elétrons fluem para o carbono 2. O dióxido de carbono deixa o carbono beta do isocitrato como um grupo de saída com os elétrons fluindo para o oxigênio cetônico do alfa C colocando uma carga negativa sobre o oxigênio do alfa C e formando uma dupla ligação alfa-beta insaturada entre os carbonos 2 e 3. O par único no oxigénio alfa-C capta um próton de um aminoácido lisina próximo.
A quarta caixa é a etapa 3, que é a saturação da dupla ligação alfa-beta insaturada entre os carbonos 2 e 3. Nesta etapa da reação, a lisina deprotona o oxigênio do carbono alfa e o par único de elétrons no oxigênio do carbono alfa desce reformando a dupla ligação cetônica e empurrando o par único (formando a dupla ligação entre o carbono alfa e beta) para fora, pegando um próton do aminoácido Tyrosine próximo. Esta reacção resulta na formação de alfa-ketoglutarato, NADH + H+/NADPH + H+, e CO2.
Mecanismo detalhadoEditar
Dois resíduos de aminoácidos de aspartato (abaixo à esquerda) estão a interagir com duas moléculas de água adjacentes (w6 e w8) no complexo Mn2+ de isocitrato de porco IDH para desprotonificar o álcool do átomo de alfa-carbono. A oxidação do alfa-C também ocorre nesta imagem onde o NAD+ aceita um hidreto resultando em oxalosuccinato. Juntamente com a mudança estereoquímica de sp3 para sp2 em torno do alfa-C, há um grupo de cetonas que é formado pelo grupo do álcool. A formação desta dupla ligação cetônica permite que a ressonância ocorra à medida que os elétrons que descem do grupo de carboxilato de saída se movem em direção à cetona.
A descarboxilação do oxalosuccinato (abaixo do centro) é um passo fundamental na formação do alfa-ketoglutarato. Nesta reação, o par solitário no grupo adjacente Tyrosine hydroxyl abstrai o próton do grupo carboxyl. Este grupo carboxil também é referido como a subunidade beta na molécula isocitrato. A desprotonificação do grupo carboxil faz com que o par único de elétrons desça fazendo dióxido de carbono e separando do oxalosuccinato. Os elétrons continuam a se mover em direção ao carbono alfa empurrando os elétrons de dupla ligação (fazendo a cetona) para abstrair um próton de um resíduo de lisina adjacente. Uma dupla ligação alfa-beta insaturada resulta entre o carbono 2 e o carbono 3. Como você pode ver na figura, o íon verde representa ou Mg2+ ou Mn2+, que é um co-fator necessário para que esta reação ocorra. O metal-íon forma um pouco complexo através de interações iônicas com os átomos de oxigênio no quarto e quinto carbonos (também conhecido como subunidade gama do isocitrato).
Após o dióxido de carbono ser separado do oxalosuccinato no passo de descarboxilação (abaixo à direita), o enol irá tautomerizar para o keto de. A formação da dupla ligação cetônica é iniciada pela desprotonificação desse oxigênio do carbono alfa (C#2) pela mesma lisina que protonou o oxigênio em primeiro lugar. O par de elétrons solitários se move para baixo dando o pontapé de saída aos pares solitários que estavam fazendo a dupla ligação. Este par solitário de elétrons abstrai um próton da Tirosina que deprotonou o grupo carboxil no passo de descarboxilação. A razão pela qual podemos dizer que os resíduos de Lys e Tyr serão os mesmos do passo anterior é porque eles estão ajudando a manter a molécula de isocitrato no local ativo da enzima. Estes dois resíduos poderão formar ligações de hidrogênio para frente e para trás desde que estejam suficientemente próximos do substrato.
 Passo Oxidoreductase onde o NAD+ é usado para aceitar um hidreto.
|
>

Descarboxilação do oxalosuccinato.
> |
 > > Saturação da dupla ligação alfa-beta insaturada.
> |
A enzima isocitrato desidrogenase como mencionado acima produz alfa-ketoglutarato, dióxido de carbono, e NADH + H+/NADPH + H+. Há três mudanças que ocorreram ao longo da reação. A oxidação do Carbono 2, a descarboxilação (perda de dióxido de carbono) do Carbono 3 e a formação de um grupo de cetonas com uma mudança estereoquímica de sp3 para sp2.
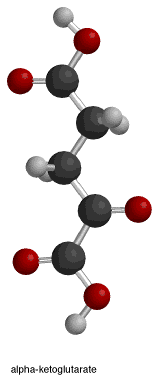 |
 NADP+ isocitrato desidrogenase dependente do porco complexado com Mn2+ e isocitrato. Vista da superfície da bolsa ativa do local onde o isocitrato é limitado por aminoácidos polares.
|
 NADP+ isocitrato dependente de isocitrato de porco complexado com Mn2+ e isocitrato.
|
>

Complexo enzimático suíno; isocitrato de sítio ativo e A.A. adjacente.
> |
Site ativoEdit
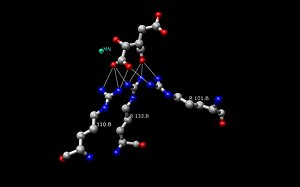 >
>A estrutura enzimática Isocitrate Dehydrogenase (IDH) na Escherichia coli foi a primeira estrutura a ser elucidada e compreendida. Desde então, a estrutura da Escherichia coli IDH tem sido usada pela maioria dos pesquisadores para fazer comparações com outras enzimas isocitrato desidrogenase. Há muito conhecimento detalhado sobre esta enzima bacteriana e descobriu-se que a maioria das desidrogenase isocitrato são semelhantes em estrutura e, portanto, também em função. Esta semelhança de estrutura e função dá uma razão para acreditar que as estruturas são conservadas, assim como os aminoácidos. Portanto, os locais ativos entre a maioria das enzimas isocitrato isocitrato desidrogenase procarióticas devem ser conservados também, o que é observado em muitos estudos feitos com enzimas procarióticas. As enzimas isocitrato isocitrato desidrogenase eucarióticas, por outro lado, ainda não foram totalmente descobertas.Cada dímero de IDH tem dois sítios ativos. Cada local ativo liga uma molécula NAD+/NADP+ e um íon metálico divalente (Mg2+,Mn2+). Em geral, cada local ativo tem uma seqüência conservada de aminoácidos para cada local de ligação específico. Em Desulfotalea psychrophila (DpIDH) e porco (PcIDH) há três substratos ligados ao local ativo.
- Isocitrato liga-se dentro do local ativo a uma seqüência conservada de cerca de oito aminoácidos através de ligações de hidrogênio. Estes ácidos incluem (podem variar em resíduos mas com propriedades similares) tirosina, serina, asparagina, arginina, arginina, arginina, tirosina, e lisina. Suas posições na espinha dorsal variam, mas estão todas dentro de uma faixa próxima (ou seja, Arg131 DpIDH e Arg133 PcIDH, Tyr138 DpIDH e Tyr140 PcIDH).
- O ião metálico (Mg2+, Mn2+) liga-se a três aminoácidos conservados através de ligações de hidrogénio. Estes aminoácidos incluem três resíduos de Aspartato.
- NAD+ e NADP+ ligam-se dentro do local ativo dentro de quatro regiões com propriedades similares entre as enzimas IDH. Estas regiões variam mas estão ao redor , , , e . Novamente as regiões variam, mas a proximidade das regiões é conservada.