Wprowadzenie
Hodowcy i badacze są zainteresowani hybrydyzacją i introgresją pomiędzy rozbieżnymi genotypami jako potencjalnymi czynnikami prowadzącymi do ekologicznej dywergencji potomstwa (Rieseberg i in., 2007; Arnold i in., 2012). I odwrotnie, hybrydyzacja może czasami wiązać się z obniżoną kondycją mieszańca, taką jak słabość i/lub sterylność w F1 i późniejszych pokoleniach, nawet w przypadku hybrydyzacji pomiędzy członkami tego samego gatunku.
Zmniejszona żywotność mieszańca i/lub płodność segregująca w F2 lub późniejszych pokoleniach jest określana jako rozpad mieszańca (HB), w którym allele recesywne są koniecznie związane. Ta bariera reprodukcyjna jest obserwowana od dawna zarówno u roślin jak i zwierząt (Dobzhansky, 1970; Grant, 1971). Wiele analiz genetycznych tej bariery ujawniło, że jest ona realizowana przez komplementarny efekt pomiędzy i/lub pomiędzy loci o zróżnicowanych allelach, powszechnie nazywany niezgodnością Batesona-Dobzhansky’ego-Mullera (BDM) (Rieseberg i Willis, 2007). HB z konieczności obejmuje wewnętrzne postzygotyczne bariery reprodukcyjne, takie jak nieżywotność mieszańca (w tym osłabienie, nekrozę i chlorozę) oraz sterylność mieszańca (w męskiej, żeńskiej lub obu gametach); dlatego niektórzy badacze mogą nie odróżniać HB od nieżywotności i sterylności u potomstwa F1. Niemniej jednak uważam, że HB jest wygodną klasyfikacją dla barier reprodukcyjnych, ponieważ implikuje ich podstawowe podstawy genetyczne (tj. zaangażowanie alleli recesywnych).
Ostatnie badania z wykorzystaniem modelowego gatunku roślin Arabidopsis zapewniły lepsze zrozumienie HB w odniesieniu do jego mechanizmów molekularnych (Vaid i Laitinen, 2019). I odwrotnie, chociaż ryż (gatunek Oryza) jest modelową rośliną uprawną, obecne zrozumienie genetycznych podstaw HB pozostaje ograniczone u tego gatunku, prawdopodobnie dlatego, że HB nie stanowi bariery reprodukcyjnej w mieszańcach F1, w których oczekuje się wyższej produkcji ziarna w porównaniu z rodzicielskimi liniami wsobnymi, i prawdopodobnie dlatego, że gorsze rośliny, które segregują w F2 i późniejszych pokoleniach, mogą być łatwo wybrane na podstawie fenotypu z populacji hodowlanej. Trudności w mapowaniu genetycznym spowodowane dziedziczeniem recesywnym mogą również stać za tym ograniczeniem.
W tym miejscu przedstawiam zarys HB w krzyżówkach ryżu, odnosząc się do informacji dostarczonych przez badania Arabidopsis i omawiam, jak HB jest traktowane w hodowli ryżu.
The Genetic Basis of Hybrid Breakdown in Rice
Uważa się, że ryż uprawny, O. sativa japonica i indica, tworzy kompleks gatunkowy ze swoim domniemanym protoplastą (O. rufipogon) i gatunkami dzikimi; jednak w krzyżowaniach między nimi obserwuje się wiele barier reprodukcyjnych, w tym HB (Oka, 1988; Vaughan i in., 2003).
Według mojej najlepszej wiedzy, Dr. Oka jako pierwszy opisał genetyczne podstawy HB w krzyżówkach międzygatunkowych między japonica i indica, co zostało wyjaśnione przez dwa komplementarne geny (Oka, 1957). Odpowiedzialne geny nie zostały jednak zmapowane na chromosomach, ponieważ markery molekularne nie były wówczas dostępne.
W mieszańcach ryżu, HB często opisywano w krzyżówkach międzyspecyficznych (O. sativa ssp. japonica × ssp. indica) i międzygatunkowych (O. sativa × O. nivara i O. sativa × O. glumaepatula) (Oka, 1957; Sato i Morishima, 1988; Wu i in…, 1995; Li et al., 1997; Fukuoka et al., 1998, 2005; Kubo i Yoshimura, 2002, 2005; Matsubara et al., 2007a,b, 2015; Yamamoto et al., 2007; Ichitani et al., 2012; dla krzyżówek międzygatunkowych; Sobrizal et al., 2001; Miura et al., 2008 dla krzyżówek międzygatunkowych) (Tabela 1). Większość z tych przypadków HB była spowodowana dwulokalną niezgodnością BDM i allelami recesywnymi, przy czym 1/16 potomstwa F2, które było homozygotyczne dla alleli recesywnych w obu loci wykazywało fenotyp HB; jednakże 4/16 potomstwa, które było heterozygotyczne tylko w jednym locus wykazywało fenotyp HB, w zależności od kombinacji krzyżowej (Tabela 1 i Rysunek 1A). W mieszańcach backcrossowych jeden locus był już utrwalony allelami pochodzącymi od rodzica rekurencyjnego; dlatego 1/4 potomstwa BCnF2 wykazywała fenotyp HB (Tabela 1 i Rysunek 1B).

Tabela 1. Genetyczne podstawy rozkładu mieszańcowego odnotowanego w krzyżówkach ryżu.
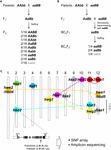
Rysunek 1. Genetyczne podstawy rozpadu mieszańców (HB). (A) Schematyczne przedstawienie genotypu HB, który segreguje w potomstwie F2. Gdy genotypami rodzicielskimi są AAbb i aaBB, 1/16 potomstwa F2 bez dominującego allelu, co jest zaznaczone szarym kolorem (np. aabb), wykazuje obniżoną żywotność i/lub płodność, chociaż pozostałe genotypy są normalne. W niektórych krzyżówkach ryżowych, potomstwo F2 z tylko jednym dominującym allelem (tj. Aabb, aaBb) również wykazuje obniżoną żywotność i/lub płodność. (B) Schematyczne przedstawienie genotypu HB, który segreguje w potomstwie BCnF2. Ponadto, 1/4 potomstwa BCnF2 wykazuje obniżoną żywotność i/lub płodność. (C) Chromosomalna lokalizacja genów leżących u podstaw HB w krzyżówkach ryżowych. Geny zostały wstępnie zmapowane w oparciu o wyniki mapowania genów w każdym z badań. Zestaw genów komplementarnych jest połączony linią przerywaną. Komplementarne geny z hbd1 i hwf1 nie zostały opisane. Przedstawiono przykład funkcjonalnego polimorfizmu nukleotydowego powodującego HB, opublikowanego przez Yamamoto i wsp. (2010), a informacje te pozwolą nam na przeprowadzenie badań metodą SNP array lub sekwencjonowania amplikonowego.
Fenotyp HB u mieszańców ryżu różni się w zależności od kombinacji krzyżowania. Na przykład, genotyp hbd2/hbd2 hbd3/hbd3 wykazuje słabość, ale bez wyraźnej sterylności nasion. Natomiast genotyp hbd4/hbd4 hbd5/hbd5 wykazuje zarówno słabość, jak i sterylność nasion (Matsubara i in., 2007a, 2015; Yamamoto i in., 2007; Tabela 1). Zdarzają się również skomplikowane przypadki, w których obserwuje się HB wykazujące zarówno słabość, jak i sterylność nasion (genotyp hwe1/hwe1 hwe2/hwe2) lub HB wykazujące wyłącznie sterylność nasion (hsa1/hsa1 hsa2/hsa2 hsa3/hsa3), pomimo tej samej kombinacji krzyżowej, tj. krzyżówki Asominori × IR24 (Kubo i Yoshimura, 2002, 2005; tab. 1). Ponieważ te HB mają niezależne podstawy genetyczne, co najmniej 5/64 potomstwa F2 wykazywałoby fenotyp HB w tej krzyżówce.
Do tej pory kilka zestawów loci odpowiedzialnych za HB zostało zmapowanych do określonych regionów genomowych przy użyciu markerów DNA w krzyżówkach pomiędzy odmianami ryżu japonica i indica (Chrs 7 i 10, Fukuoka i in., 1998; Chrs 1 i 12, Wu i in., 1995; Kubo i Yoshimura, 2002; Matsubara i in., 2015; Chrs 6 i 11, Fukuoka i in., 2005; Chrs 8, 9 i 12, Kubo i Yoshimura, 2005; Chrs 2 i 11, Matsubara i in., 2007a; Yamamoto i in., 2007, 2010; Chrs 11 i 12, Ichitani i in., 2012; Tabela 1 i Rysunek 1C). Wyniki tych badań wykazały, że loci leżące u podstaw HB ryżu są wspólne w niektórych krzyżówkach, ale różnią się w innych, co sugeruje ich zróżnicowanie w genomach ryżu. Na przykład, w niektórych przypadkach allel HB przenoszony przez O. nivara, gatunek blisko spokrewniony z odmianami incica, dzieli ten sam locus z odmianą indica; natomiast allel HB przenoszony przez O. glumaepatula, gatunek blisko spokrewniony z odmianami uprawnymi, nie dzieli locus z żadną inną odmianą (Tabela 1 i Rysunek 1C). Należy zauważyć, że opisane powyżej przypadki HB są spowodowane przez zestaw genów o głównym wpływie; jednakże istnieją również przypadki spowodowane przez zestaw genów o mniejszym wpływie, np. nieznacznie obniżona płodność nasion, których nie można pominąć w hodowli ryżu, a które nie zostały jeszcze wykryte.
Molecular Mechanisms Underlying Hybrid Breakdown
Odpowiedź autoimmunologiczna: Bomblies i wsp. (2007) po raz pierwszy donieśli, że odpowiedź autoimmunologiczna, że gen odporności na choroby NB-LRR lub gen R jest związany z Arabidopsis HB w krzyżówkach wewnątrzgatunkowych, chociaż ta odpowiedź autoimmunologiczna jest głównie wyrażona jako nekroza w roślinach F1, a HB w potomstwie F2 wydawała się być uwarunkowana temperaturą. U mieszańców Arabidopsis opisano kilka linii dowodowych na występowanie HB spowodowanego reakcją autoimmunologiczną (Alcázar i in., 2009, 2010). Zaangażowanie genów NB-LRR w HB sugeruje, że wiele regionów genomowych może być związanych z Arabidopsis HB; w rzeczywistości obszerne badanie przeprowadzone przez Chae i wsp. (2014) wspiera tę ideę.
Także w hybrydach ryżu, HB spowodowane przez odpowiedź autoimmunologiczną zostało zgłoszone w krzyżu japonica × indica (Yamamoto i wsp., 2010). W tym przypadku HB występuje u roślin F2, gdy gen hybrid breakdown 2 (hbd2), który koduje kinazę kazeinową I i jest przenoszony przez odmianę indica, jest połączony z grupą genów R przenoszonych przez odmianę japonica. Ponieważ te allele przyczynowe prawdopodobnie zachowują się jak allele częściowo recesywne, w grę mogą wchodzić produkty genowe (Matsubara i in., 2007a; Yamamoto i in., 2007, 2010).
Reciprocal silencing of duplicated genes: pojęcie to zostało po raz pierwszy teoretycznie zaproponowane jako genetyczna podstawa niezgodności mieszańcowej (Werth i Windham, 1991; Lynch i Force, 2000). W modelu tym jedna populacja traci funkcję w jednym locus, a zachowuje ją w drugim, podczas gdy druga populacja doświadcza odwrotnego efektu. W konsekwencji, 1/16 zygot F2 nie posiada funkcjonalnych genów (Lynch i Force, 2000). Dobrym przykładem tego zjawiska jest HB u mieszańców Arabidopsis opisany przez Bikard et al. (2009) oraz Vlad et al. (2010). W tym scenariuszu odnotowano również przypadki obejmujące wyciszanie epigenetyczne (Agorio i in., 2017; Blevins i in., 2017).
Jako że ryż doświadczył zarówno duplikacji całogenomowej, jak i segmentowej (Wang i in., 2007; Guo i in., 2019), wzajemne wyciszanie może być powszechne jako przyczyna niezgodności mieszańcowej. Jednak do tej pory nie ma dowodów na HB spowodowane wzajemnym wyciszaniem zduplikowanych genów u ryżu.
Niezależnie od odpowiedzi autoimmunologicznej i wzajemnego wyciszania, zidentyfikowano interakcję pomiędzy helikazą 18 DEAD-box RNA i innym genem (MORPHEUS MOLECULE 1 jako prawdopodobny kandydat), które powodują HB u mieszańców Arabidopsis (Plötner i in., 2017; Vaid i in., 2020). Co ciekawe, fenotyp HB zmniejsza się między pokoleniami F3 i F4, sugerując epigenetyczną regulację ekspresji genów.
W mieszańcach ryżu, wśród trzech loci sterylności hybrydowej (hsa), Kubo i wsp. (2016) wykazali ostatnio, że locus hsa1 składa się z dwóch genów, i że geny te kodują odpowiednio białko DUF1618 i niescharakteryzowane białko z pewnym podobieństwem do białka wiążącego nukleotydy. Cechy molekularne pozostałych genów komplementarnych, hsa2 i hsa3, nie zostały opisane.
Połamanie hybrydowe w konwencjonalnej hodowli ryżu
Przez dziesięciolecia metoda bulk-population była szeroko stosowana w konwencjonalnej hodowli ryżu. W tej metodzie, po skrzyżowaniu, populacja F4 lub F5 jest hodowana przez samozapłodnienie luzem (a nie metodą zstępowania pojedynczych nasion) bez sztucznej selekcji, ale populacja wczesnego pokolenia jest poddawana selekcji naturalnej i żywotności (Allard, 1960; Ikehashi i Fujimaki, 1980 dla szczegółów). W metodzie bulk-population można się spodziewać, że wiele słabych i/lub sterylnych genotypów zostanie wyeliminowanych z populacji przed utworzeniem populacji zaawansowanego pokolenia; dlatego metoda ta może dostarczyć ograniczonych informacji o HB.
W celu introgresji lub akumulacji pożądanych cech od dawcy(ów) do odmiany, hodowcy ryżu często przeprowadzali krzyżowanie wsteczne lub krzyżowanie wielu rodziców, a następnie metodę bulk-population. Empirycznie od dawna wiadomo, że te metody krzyżowania pozwalają na złagodzenie utraty kandydatów do selekcji przez bariery reprodukcyjne, ponieważ metody te często ograniczają segregację destrukcyjnych kombinacji alleli związanych z HB u potomstwa mieszańca. Jednakże łagodzenie barier reprodukcyjnych przez te metody krzyżowania jest nieuchronnie zależne od genotypu HB rodziców.
Hybrid Breakdown in Genomics-Assited Breeding
Publikacja referencyjnych sekwencji genomów roślin uprawnych oraz rozwój technologii sekwencjonowania następnej generacji przyspieszyły postęp w hodowli molekularnej roślin uprawnych (Kole i in., 2015). W tym kontekście hodowla wspomagana genomiką, taka jak selekcja genomowa oparta na genotypach markerów DNA o zasięgu genomowym, została rozważona w hodowli roślin uprawnych (Spindel i Iwata, 2018).
W hodowli wspomaganej genomiką roślin samopylnych populacje zaawansowanej generacji, takie jak rekombinowane linie wsobne, są często wykorzystywane jako populacje referencyjne, z których uzyskuje się dane genotypowe i fenotypowe o zasięgu genomowym. Następnie u potomstwa wczesnych pokoleń (np. F2) prowadzi się selekcję opartą wyłącznie na genotypach markerowych. W tym schemacie selekcji należy zauważyć, że populacja referencyjna zwykle nie dostarcza informacji o regionie genomowym dla HB, ponieważ słabe i/lub sterylne potomstwo powinno być wyeliminowane we wczesnych pokoleniach po skrzyżowaniu. Dlatego też, jeśli przyjmiemy taki schemat selekcji, będziemy musieli zrezygnować z niektórych ważnych kandydatów do selekcji. Alternatywnie, możemy wybrać niepożądanych kandydatów tak, że HB ujawni się w późniejszych pokoleniach.
Dyskusja
Pomimo ograniczonej liczby badań na ten temat, wydaje się, że loci leżące u podstaw HB w krzyżowaniach ryżu są raczej zróżnicowane niż wspólne (Tabela 1 i Rysunek 1). Jak opisano powyżej, informacja o dystrybucji alleli związanych z HB wśród rodziców krzyżówki powinna być warunkiem wstępnym dla hodowli ryżu, w szczególności dla maksymalizacji efektywności hodowli wspomaganej genomiką. Dlatego konieczne jest dodatkowe wykrywanie HB w krzyżówkach ryżowych i mapowanie odpowiedzialnych za to genów w genomie ryżu. W mieszańcach Arabidopsis takie dane o genach R i genach z nimi współdziałających zostały szeroko przebadane (Alcázar i in., 2010, 2014; Chae i in., 2014). Raportowanie genu hbd2 przez Yamamoto i wsp. (2010) w hybrydzie ryżowej jest dobrym przykładem obszernego badania wykonanego przy użyciu tablic SNP lub sekwencjonowania amplikonowego (Rysunek 1C). Geny hbd3 i hsa1 są również kandydatami do tego typu badań. Ostatecznie, opracowanie kryteriów, które pozwolą nam przewidzieć HB na podstawie informacji genomowych linii rodzicielskich, posłuży jako ważne narzędzie dla hodowli wspomaganej genomiką. Nawet jeśli czynnik sprawczy nie został zidentyfikowany jako pojedynczy gen, blisko powiązane markery, takie jak polimorfizmy pojedynczych nukleotydów, będą skuteczne w klasyfikowaniu haplotypów związanych z HB, ponieważ szacuje się, że nierównowaga powiązań ryżu uprawnego sięga 100-200 kb, podczas gdy u dzikich gatunków (takich jak O. rufipogon) może rozciągać się na kilkadziesiąt kb (Mather i in., 2007; McNally i in., 2009; Huang i in., 2010). Informacje te umożliwią zaprojektowanie bardziej wydajnej i skutecznej kombinacji krzyżowej.
Bulked segregant analysis followed by next-generation sequencing can be useful for the mapping of HB-associated loci, as well as conventional linkage mapping (such as quantitative trait locus analysis), because HB segregants often show distinguishable features (about traits such as plant height, tiller number, and fertility) from normal growth segregants in each cross population. Pozyskiwanie danych obrazowych przy użyciu kamery cyfrowej i drona może również odegrać ważną rolę w fenotypowaniu w laboratorium i na polu ryżowym. Te wysiłki dostarczą cennych informacji nie tylko hodowcom ryżu, ale także biologom ewolucyjnym.
Co więcej, jeśli allel związany z HB zostanie zidentyfikowany, zaburzenie genu poprzez wiązkę jonów lub edycję genomu może pomóc przezwyciężyć HB, czego przykładem jest sterylność hybryd F1 w ryżu (Koide i in., 2018; Xie i in., 2019).
Data Availability Statement
Wszystkie zestawy danych przedstawione w tym badaniu są zawarte w artykule/materiale uzupełniające.
Author Contributions
Autor potwierdza, że jest jedynym współautorem tej pracy i zatwierdził ją do publikacji.
Konflikt interesów
Autorzy deklarują, że badania zostały przeprowadzone przy braku jakichkolwiek komercyjnych lub finansowych relacji, które mogłyby być interpretowane jako potencjalny konflikt interesów.
Podziękowania
Chciałbym zadedykować tę pracę pamięci Dr. Darshana Brara. Był on wybitnym hodowcą ryżu, który energicznie wprowadzał użyteczne geny przenoszone przez dzikie gatunki do odmian ponad barierami reprodukcyjnymi.
Allard, R. W. (1960). Principles of Plant Breeding. New York, NY: John Wily and Sons, Inc.
Google Scholar
Dobzhansky, T. (1970). Genetyka procesu ewolucyjnego. New York, NY: Columbia University Press.
Google Scholar
Grant, V. (1971). Plant Speciation. New York, NY: Columbia University Press.
Google Scholar
Ikehashi, H., and Fujimaki, H. (1980). „Modified bulk population method for rice breeding,” in Proceedings of the International Rice Research Conference on Innovative approaches to rice breeding, (Los Baños, PA: International Rice Research Institute), 163-182.
Google Scholar
Oka, H.-I. (1988). Origin of Cultivated Rice. Tokyo: JSSP.
Google Scholar
.