Wprowadzenie: Defining Terms
Wcześniejsze dane dotyczące taksonomii Cannabis zostały wcześniej przejrzane (Russo, 2007), które zostaną tutaj streszczone i uzupełnione. Cannabis to dwupienny jednoroczny gatunek należący do rodziny Cannabaceae, do której tradycyjnie zalicza się chmiel, Humulus spp. Alternatywnie, na podstawie map miejsc restrykcyjnych chloroplastu (Weigreffe i in., 1998) oraz sekwencji chloroplastowego genu mat K (Song i in., 2001), Cannabis przypisano także do rodzin Moraceae, Urticaceae, a nawet Celtidaceae. Ostatnio do Cannabaceae zaliczono osiem rodzajów: Celetis, Pteroceltis, Aphananthe, Chaetachme, Gironniera, Lozanella, Trema i Parasponia, obejmujących 170 nieparzystych gatunków (McPartland, 2018), co zostało poparte analizą genetyczną czterech loci plastydowych (Yang i in., 2013). Obecne badania kopalnych próbek pyłku związane z ekologicznymi związkami Cannabis z towarzyszącymi gatunkami stepowymi (Poaceae, Artemisia, Chenopodiaceae), a Humulus (chmiel) z rodzajami leśnymi (Alnus, Salix, Populus), ustaliły, że chociaż Cannabis wydaje się pochodzić z Płaskowyżu Tybetańskiego co najmniej 19,6 miliona lat temu, był również autochtoniczny w Europie od co najmniej miliona lat (McPartland i in., 2018), i obalił konwencjonalną mądrość, że ten „zwolennik obozu” został przyniesiony tam przez człowieka.
Samo przypisanie gatunkowe Cannabis jest obarczone wielką debatą. Cannabis sativa, co oznacza „uprawiane Cannabis”, został tak nazwany przez Fuchsa, między innymi, w 1542 roku (Fuchs, 1999), przypisanie 211 lat przed systematyzacji botanicznych binomials Linnaeus w jego Species Plantarum (Linnaeus, 1753). Lamarck zasugerował następnie Cannabis indica, bardziej zdrobniałą odurzającą roślinę indyjską z Indii, jako oddzielny gatunek (Lamarck, 1783). Kwestia ta pozostała nierozstrzygnięta w kolejnych stuleciach, z dwoma przeciwstawnymi filozofiami. Ernest Small był orędownikiem koncepcji jednego gatunku (Small i Cronquist, 1976). Poliptypowe traktowanie Cannabis również zyskało zwolenników (Schultes i in., 1974; Anderson, 1980) na podstawie kryteriów morfologicznych sugerujących rozdzielenie Cannabis sativa L. Cannabis indica Lam. i Cannabis ruderalis Jan., schemat wspierany przez systematyczną chemotaksonomię. Analiza składowych głównych (PCA) 157 dostępów Cannabis z całego świata, w której oceniono częstotliwości allozymów w 17 loci genowych, sugeruje podział (Hillig, 2005b). Pule genów „sativa” z próbek ruderalnych z Europy Wschodniej były powiązane z wąskolistnymi europejskimi i środkowoazjatyckimi roślinami włóknistymi i nasiennymi, podczas gdy grupa „indica” obejmowała dalekowschodnie rośliny nasienne i włókniste oraz rośliny narkotyczne o szerokich liściach z większości pozostałych części świata, wraz z dzikimi dostępami z subkontynentu indyjskiego. Uważano, że próbki z Azji Środkowej (Cannabis ruderalis) reprezentują trzecią grupę. Badania chromatografii gazowej (GC) i elektroforezy w żelu skrobiowym również sugerowały rozdzielenie gatunkowe sativa i indica (Hillig i Mahlberg, 2004).
Czynniki agronomiczne w 69 próbkach sugerowały włączenie wschodnich konopi i roślin narkotycznych do Cannabis indica (Hillig, 2005a), podział poparty polimorfizmem długości fragmentów (Datwyler i Weiblen, 2006).
W ostatnim czasie PCA zdawała się wskazywać na zawartość terpenoidów jako najbardziej przekonujące markery rozróżniające chemotaksonomiczne pomiędzy gatunkami putative sativa i indica (Elzinga i in., 2015). Podobnie, PCA była odczuwana do oddzielenia narkotycznych Cannabis od konopi (Sawler et al., 2015). Ostatnie badania wykazały demarkację Cannabis narkotycznych od dostępów konopi poprzez genotypowanie 13 mikrosatelitarnych loci w całym genomie, a nie tylko genów wpływających na produkcję kannabinoidów lub włókien (Dufresnes i in., 2017). Profesor Giovanni Appendino zgłosił obecność stereoizomeru cis-Δ9-THC tylko w dostępach konopi (Giovanni Appendino, komunikacja osobista). Jednak te rozróżnienia mogą zostać pominięte, biorąc pod uwagę obecną tendencję do krzyżowania konopi z odmianami narkotykowymi w celu uniknięcia ograniczeń prawnych dotyczących zawartości THC.
Kontrowersje dotyczące gatunków konopi, Cannabis sativa vs. indica vs. afghanica, trwają nieprzerwanie do dnia dzisiejszego, z beznamiętnymi argumentami wysuwanymi przez protagonistów (Clarke i Merlin, 2013, 2016; Small, 2015; McPartland i Guy, 2017; Small, 2017). Ten autor, będąc po każdej stronie problemu w tym czy innym czasie, zdecydował się uciec od niemożliwej do pogodzenia debaty taksonomicznej jako niepotrzebnego rozproszenia uwagi (Piomelli i Russo, 2016) i raczej podkreślić, że istotne są tylko biochemiczne i farmakologiczne rozróżnienia pomiędzy dostępami Cannabis. W swoim ostatnim przeglądzie McPartland zgodził się, że „kategoryzowanie Cannabis jako 'Sativa’ i 'Indica’ stało się ćwiczeniem bezcelowym. Wszechobecne krzyżowanie i hybrydyzacja sprawiają, że ich rozróżnienie jest bez znaczenia.” (McPartland, 2018) (s. 210).
Dodatkowa nie-sensowna kontrowersja nomenklaturowa odnosi się w potocznym języku do „szczepów” Cannabis, określenia, które jest odpowiednie dla bakterii i wirusów, ale nie roślin (Bailey i Bailey, 1976; Usher, 1996; Brickell i in., 2009), szczególnie tak jest w przypadku Cannabis, gdzie odmiana chemiczna, skrót „chemovar” jest najbardziej odpowiednim określeniem (Lewis i in., 2018).
Genom Cannabis i alternatywna produkcja biochemiczna gospodarza
2011 był przełomowym rokiem dla genomiki Cannabis, ponieważ Medical Genomics i Nimbus Informatics wydały raport online na temat kompletnej sekwencji genomowej 400 milionów par bazowych, do której wkrótce dołączył projekt genomu i transkryptomu (van Bakel i in., 2011).
Ten rozwój wywołał znaczący rozgłos i kontrowersje, co może zapowiadać. Podczas gdy ludzki genom został przeanalizowany około 20 lat wcześniej, implikacje dla Cannabis były przedmiotem wielkich spekulacji.
Wiadomości te stały się katalizatorem gorączki nowych badań, ale znaczny postęp został już osiągnięty w genetyce stosowanej Cannabis. Identyfikacji i syntezy Δ9-tetrahydrokannabinolu (THC) dokonano w Izraelu w 1964 r. (Gaoni i Mechoulam, 1964), ale dopiero znacznie później udało się sklonować enzym biosyntezy, syntazę kwasu tetrahydrokannabinolowego (syntaza THCA) (Sirikantaramas i in., 2004; Rysunek 1). Następnie przeprowadzono krystalizację enzymu (Shoyama i in., 2005). Syntaza kwasu kannabidiolowego, która katalizuje kwas kannabidiolowy (CBDA), prekursor kannabidiolu (CBD), została wcześniej zidentyfikowana i wyprodukowana w czystej postaci (Taura i in., 1996; Rysunek 1). Te wydarzenia pobudziły dodatkowe odkrycia, w tym archeologiczne odkrycie fitochemiczne syntazy THCA w liczącej 2700 lat skrytce konopi z grobowca w Azji Środkowej wraz z dwoma wcześniej niezgłaszanymi polimorfizmami pojedynczego nukleotydu (SNP) w sekwencji genu enzymu (Russo i in., 2008).
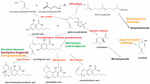
Rysunek 1. Ścieżki biosyntezy i enzymy (kolor czerwony) Cannabis sativa, wskazanie gatunku naturalnego Helichrysum umbraculigerum oraz gatunków alternatywnych (kolor), które zostały zmodyfikowane genetycznie w celu wytworzenia kolejnych produktów.
Do 2011 roku zidentyfikowano enzymy do produkcji głównych fitokannabinoidów. Podobnie, selektywna zaawansowana hodowla mendlowska dała odmiany Cannabis bogate w konkretne pojedyncze składniki. W ten sposób wyprodukowano rośliny o wysokiej zawartości THC i CBD do celów rozwoju farmaceutycznego (de Meijer i in., 2003; de Meijer, 2004), z analogiczną hodowlą linii o wysokiej zawartości kannabigerolu (CBG) (de Meijer i Hammond, 2005) i kannabichromenu (CBC) (de Meijer i in., 2009a). Selektywna hodowla objęła również analogi propylowych fitokannabinoidów, tetrahydrokannabiwarynę (THCV), kannabidiwarynę (CBDV), kannabigeriwarynę (CBGV) i kannabichromiwarynę (CBCV) (de Meijer, 2004). Dostępność roślin o wysokim mianie tych „pomniejszych kannabinoidów” zapowiada nowe interesujące zastosowania farmaceutyczne (Russo, 2011; Russo i Marcu, 2017).
Dostęp do genomu Cannabis może uprościć produkcję roślin pozbawionych THC za pomocą technologii CRISPR (clustered regularly-interspaced short palindromic repeats). Choć mogłoby to być atrakcyjne dla hodowli konopi przemysłowych, już wcześniejsze pokolenie hodowców roślin dało odmiany konopi, które z łatwością spełniają międzynarodowe restrykcje wymagające zawartości THC na poziomie 0,1% lub mniejszym (Wirtshafter, 1997; McPartland i in., 2000; Small i Marcus, 2003). W rzeczywistości, Cannabis bez kannabinoidów, bez funkcjonalnej syntazy kwasu kannabigerolowego (Rysunek 1) również został wyprodukowany w sposób konwencjonalny (de Meijer i in., 2009b). Nie jest więc jasne, czy inżynieria genetyczna konopi jest w ogóle konieczna w przypadku tej rośliny, której niezwykła plastyczność już wykazuje ogromną różnorodność biochemiczną. Wprowadzenie genetycznie zmodyfikowanych organizmów (GMO) Cannabis wywołałoby znaczne kontrowersje wśród niektórych segmentów populacji i prawdopodobnie sprowokowałoby lawinę prawnych uwikłań dotyczących praw patentowych i hodowlanych.
Można sobie łatwo wyobrazić różne dodatkowe scenariusze science fiction. W latach 90. w Internecie krążyła plotka, że apokryficzny profesor Nanofsky wprowadził geny do produkcji THC do pomarańczy (Citrus x. sinensis (L.) Osbeck). Mimo, że jest to technologicznie osiągalne, taki wysiłek byłby niczym więcej niż laboratoryjnym karnawałem w świetle niesamowitej produkcji kannabinoidów z samych konopi. Bardziej logicznym wyborem dla takich podziemnych, wywrotowych mrzonek może być ukradkowy chemowar mięty pieprzowej (Menthax piperita Lamiaceae), który zawiera nielegalne fitokannabinoidy w gruczołowych włośnikach swoich liści i wysyła kłącza i rozłogi wzdłuż cieków wodnych na całym świecie.
Pierwsze twierdzenia o produkcji kannabidiolu z chmielu (Humulus lupulus L. Cannabaceae) i lnu (Linum usitatissimum L. Linaceae) są nieuzasadnione, ale kwas kannabigerolowy i kannabigerol zostały wykryte w południowoafrykańskim Helichrysum umbraculigerum Less. Asteraceae (Bohlmann i Hoffmann, 1979; Appendino et al., 2015; Russo, 2016; Rysunek 1), ale bez odniesienia do jego stężenia. To twierdzenie zostało ostatnio potwierdzone śladowymi ilościami obserwowanymi z wysuszonych próbek części powietrznych (Mark Lewis, komunikacja osobista).
Ponieważ złożoność czysto de novo biochemicznej syntezy kannabinoidów została uznana za nieopłacalną (Carvalho i in., 2017), zaproponowano alternatywne gospodarze mikrobiologiczne (Zirpel i in., 2017). W 2004 roku udało się sklonować cDNA syntazy THCA, umożliwiającej konwersję kwasu kannabigerolowego (CBGA) do THC (Sirikantaramas i in., 2004), a także wykazano 8% produkcję THCA w korzeniach włochatych tytoniu (Nicotiana tabacum cv.Xanthi Solanaceae) po podaniu CBGA (Rysunek 1). Enzym uległ również ekspresji w owadzie Spodoptera frugiperda (J.E. Smith) Noctuidae (mól jesienny) poprzez rekombinowany bakulowirus. Następnie grupa badawcza zwróciła się w kierunku drożdży Pichia pastoris (obecnie Komagataella phaffii Phaff Saccharomycetaceae) (Taura i in., 2007; Rysunek 1) i osiągnęła konwersję CBGA do THCA na poziomie 98% w ciągu 24 godzin, z wydajnością 32,6 mg/L pożywki. Rekombinowana forma syntazy THCA okazała się 4,5-krotnie wydajniejsza niż u Cannabis i 12-krotnie wydajniejsza niż u S. frugiperda. Proces ten został następnie zoptymalizowany z 64,5-krotną poprawą aktywności (Zirpel et al., 2018), z odnotowaną produkcją w K. phaffii 3,05 g/L THCA po 8 h inkubacji w 37°C. Prosta kalkulacja zapewnia, że taka wydajność mogłaby być również osiągnięta z ekstrakcji zaledwie 15 g 20% THCA ziołowego Cannabis.
Produkcja terpenoidów konopi jest podobnie możliwa w alternatywnych gospodarzach. Mutanty Saccharomyces cerevisiae Meyen ex E.C. Hansen Saccharomycetaceae z niedoborem enzymu syntazy difosforanu farnezylu gromadzą zamiast niego pirofosforan geranylu, który jest kierowany do produkcji medycznie użytecznego terpenoidu, linalolu (Oswald et al., 2007; Rysunek 1). Podobnie, inni badacze wykorzystali biosyntetyczne możliwości mitochondriów w S. cerevisiae do zwiększenia produkcji seskwiterpenoidów z difosforanu farnezylu (Farhi i in., 2011), choć nie tych wspólnych dla Cannabis.
Obecnie, istniejące sekwencje genomowe Cannabis nie są w pełni zanotowane. W związku z tym, aby uzyskać praktyczne dane na temat funkcji genetycznych w Cannabis, konieczne będzie zastosowanie wiedzy wstępnej i pracy detektywistycznej. Największy potencjał w takich badaniach będzie tkwił w sferze epigenetyki, leżącej u podstaw dziedzicznych zmian w ekspresji genów lub fenotypie rośliny. Najbardziej odczuwalnym brakiem jest brak wiedzy na temat regulacji produkcji kannabinoidów. Zrozumienie ścieżek biosyntezy i regulacji syntaz terpenowych produkujących terpenoidy Cannabis zostało ledwie zapoczątkowane (Booth i in., 2017) i pozostaje dojrzałym celem dodatkowych badań (Russo, 2011).
Dodatkowym problemem w hodowli Cannabis pozostaje brak okazów voucherowych (które są zabronione przez US Drug Enforcement Administration bez licencji Schedule I) i formalnych depozytów akcesji chemovar w repozytoriach nasion i tkanek. To ostatnie zostało zrealizowane przez GW Pharmaceuticals oraz niezależnie przez NaPro Research (Lewis i in., 2018) w National Collection of Industrial, Food and Marine Bacteria (NCIMB) w Szkocji. Wiele prywatnych firm unikało dzielenia się germlazmą ze względu na ograniczenia prawne i strach przed utratą własności intelektualnej.
Synergia konopi
W 1998 roku profesorowie Raphael Mechoulam i Shimon Ben-Shabat stwierdzili, że układ endokannabinoidowy wykazuje „efekt świty”, w którym różnorodne „nieaktywne” metabolity i ściśle powiązane cząsteczki znacznie zwiększają aktywność podstawowych endogennych kannabinoidów, anandamidu i 2-arachidonoyloglicerolu (Ben-Shabat i in., 1998). Postulowali oni również, że pomaga to wyjaśnić, dlaczego leki botaniczne są często bardziej skuteczne niż ich wyizolowane składniki (Mechoulam i Ben-Shabat, 1999). Chociaż synteza pojedynczej cząsteczki pozostaje dominującym modelem rozwoju farmaceutyków (Bonn-Miller i in., 2018), równolegle obszernie przedstawiono koncepcję synergii botanicznej, powołując się na farmakologiczny wkład „pomniejszych kannabinoidów” i terpenoidów Cannabis w ogólny efekt farmakologiczny rośliny (McPartland i Pruitt, 1999; McPartland i Mediavilla, 2001; McPartland i Russo, 2001, 2014; Russo i McPartland, 2003; Wilkinson i in., 2003; Russo, 2011). Ilustracją jest kilka trafnych przykładów efektu świty w konopiach indyjskich:
W randomizowanym, kontrolowanym badaniu z zastosowaniem oromukosowych ekstraktów na bazie konopi indyjskich u pacjentów z nieprzezwyciężonym bólem pomimo zoptymalizowanego leczenia opioidami, ekstrakt z przewagą THC nie odróżniał się korzystnie od placebo, natomiast ekstrakt z całej rośliny (nabiximols, vide infra) zawierający zarówno THC, jak i kannabidiol (CBD) okazał się statystycznie istotnie lepszy od obu tych substancji (Johnson i in., 2010), przy czym jedyną istotną różnicą była obecność CBD w tym ostatnim.
W badaniach analgezji na zwierzętach, czyste CBD wytwarza dwufazową krzywą dawka-odpowiedź, taką, że mniejsze dawki zmniejszają reakcje bólowe, aż do osiągnięcia szczytu, po którym dalsze zwiększanie dawki jest nieskuteczne. Co ciekawe, zastosowanie ekstraktu Cannabis o pełnym spektrum działania z równoważnymi dawkami CBD eliminuje odpowiedź dwufazową na korzyść liniowej krzywej dawka-odpowiedź, tak że ekstrakt botaniczny działa przeciwbólowo w każdej dawce bez obserwowanego efektu pułapowego (Gallily i in., 2014).
Niedawne badanie kilku ludzkich linii komórkowych raka piersi w hodowli i wszczepionych guzach wykazało wyższość leczenia ekstraktem z konopi indyjskich nad czystym THC, najwyraźniej przypisywaną w tym pierwszym przypadku obecności niewielkich stężeń kannabigerolu (CBG) i kwasu tetrahydrokannabinolowego (THCA) (Blasco-Benito i in., 2018).
Antykonwulsyjne działanie kannabidiolu zauważono u zwierząt w latach 70. XX wieku, przy czym pierwsze próby na ludziach przeprowadzono w 1980 roku (Cunha i in., 1980). W niedawnym eksperymencie na myszach z napadami wywołanymi przez pentylenetetrazol zastosowano pięć różnych ekstraktów Cannabis o równych stężeniach CBD (Berman i in., 2018). Chociaż wszystkie ekstrakty wykazały korzyści w porównaniu z nieleczonymi kontrolami, zaobserwowano istotne różnice w profilach biochemicznych kannabinoidów innych niż CBD, co z kolei doprowadziło do znaczących różnic w liczbie myszy, u których wystąpiły napady toniczno-kloniczne (21,5-66,7%) i przeżywalności (85-100%), podkreślając znaczenie tych „drobnych” składników. Badanie to podkreśla konieczność standaryzacji w rozwoju farmaceutycznym, i chociaż może być interpretowane jako wsparcie modelu terapeutycznego pojedynczej cząsteczki (Bonn-Miller i in., 2018), wymaga podkreślenia, że złożone substancje botaniczne mogą spełniać amerykańskie standardy FDA (Food and Drug Administration, 2015). Konkretnie, dwa leki na bazie Cannabis osiągnęły zatwierdzenie regulacyjne, Sativex®(nabiximols, US Adopted Name) w 30 krajach i Epidiolex® w Stanach Zjednoczonych.
Wtedy pojawia się pytanie: Czy preparat Cannabis lub pojedyncza cząsteczka może być zbyt czysta, zmniejszając tym samym potencjał synergistyczny? Najnowsze dane potwierdzają, że jest to wyraźna możliwość. Anegdotyczne informacje od klinicystów wykorzystujących ekstrakty Cannabis o wysokiej zawartości CBD w leczeniu ciężkiej padaczki, takiej jak zespoły Dravet i Lennox-Gastaut, wykazały, że ich pacjenci wykazywali znaczną poprawę w zakresie częstotliwości napadów (Goldstein, 2016; Russo, 2017; Sulak i in., 2017) przy dawkach znacznie niższych niż te zgłaszane w formalnych badaniach klinicznych Epidiolexu, preparatu CBD o czystości 97% z usuniętym THC (Devinsky i in., 2016, 2017, 2018; Thiele i in., 2018). Obserwacja ta została niedawno poddana metaanalizie 11 badań obejmujących łącznie 670 pacjentów (Pamplona i wsp., 2018). Wyniki te wykazały, że u 71% pacjentów nastąpiła poprawa po zastosowaniu ekstraktów Cannabis z przewagą CBD vs. 36% na oczyszczonym CBD (p < 0,0001). Wskaźnik odpowiedzi przy 50% poprawie częstości napadów nie różnił się statystycznie w obu grupach, a obie grupy osiągnęły status wolny od napadów u około 10% pacjentów. Jednakże średnie dawki dobowe były znacząco rozbieżne w grupach: 27,1 mg/kg/d dla oczyszczonego CBD vs. tylko 6,1 mg/kg/d. dla bogatych w CBD ekstraktów Cannabis, dawka tylko 22,5% dawki dla samego CBD. Co więcej, częstość występowania łagodnych i ciężkich zdarzeń niepożądanych była ewidentnie wyższa u pacjentów z oczyszczonym CBD vs. z ekstraktem o wysokiej zawartości CBD (p < 0,0001), co autorzy przypisali stosowanej niższej dawce, która ich zdaniem została osiągnięta dzięki synergistycznemu wkładowi innych związków towarzyszących. Takie obserwacje wspierają hipotezę o większej skuteczności ekstraktów Cannabis łączących wiele składników przeciwdrgawkowych, takich jak CBD, THC, THCA, THCV, CBDV, linalool, a nawet kariofilen (Lewis i in., 2018).
Te badania i inne dostarczają solidnych podstaw dla synergii Cannabis, a także wsparcia dla rozwoju leków botanicznych w porównaniu z rozwojem pojedynczych składników (Bonn-Miller i in., 2018), lub produkcji za pomocą metod fermentacji w drożdżach lub innych mikroorganizmach. Przykład mocy konwencjonalnej hodowli selektywnej jest zilustrowany (Ryc. 2), w postaci chemowaru Cannabis nazwanego CaryodiolTM ze względu na zwiększoną zawartość kariofilenu (0,83%) jako agonisty CB2, wraz z wysoce korzystnym stosunkiem THC:CBD typu III wynoszącym 1:39,4. Taki preparat ma szansę znaleźć zastosowanie w leczeniu wielu schorzeń klinicznych, w tym: bólu, stanów zapalnych, zaburzeń włóknienia, uzależnień, lęków, depresji, chorób autoimmunologicznych, schorzeń dermatologicznych i nowotworów (Pacher i Mechoulam, 2011; Russo, 2011; Xi i in., 2011; Russo i Marcu, 2017; Lewis i in., 2018). Wytworzenie takiej kombinacji ze źródeł mikrobiologicznych mogłoby wymagać połączenia kannabinoidów pochodzących z wielu gatunków drożdży i w rezultacie stanowiłoby produkt kombinowany podlegający trudnej ścieżce regulacyjnej w porównaniu z preparatami Cannabis z ekstraktów jednego gatunku (np. nabiximols), który został zaakceptowany jako preparat jednostkowy w 30 krajach na całym świecie (Food and Drug Administration, 2015).
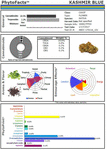
Figure 2. Przedstawienie PhytoFactsTM zawartości kannabinoidów i terpenoidów w CaryodiolTM, aka „Kashmir Blue”, chemovar typu III, z przewagą kannabidiolu i kariofylenu. Zobacz (Lewis et al., 2018), aby poznać szczegóły PhytoFacts i metodologię hodowli konwencjonalnej. Copyright© 2016 BHC Group, LLC. Wszelkie prawa zastrzeżone. Wszelkie nieautoryzowane wykorzystanie tego dokumentu lub obrazów lub znaków powyżej może naruszać prawa autorskie, znaki towarowe i inne obowiązujące przepisy.
W niniejszym artykule krótko nakreślono ostatnio technologiczne próby „ponownego wynalezienia koła fitokannabinoidowego”. Uzasadnione argumenty przemawiają za tym, że można to zrobić, ale czy powinno się to zrobić? Dane potwierdzające istnienie synergii Cannabis i zdumiewająca plastyczność genomu Cannabis sugerują rzeczywistość, która wyklucza potrzebę alternatywnych gospodarzy, a nawet inżynierii genetycznej Cannabis sativa, udowadniając w ten sposób, że „roślina robi to lepiej.”
Wkład autora
Autor potwierdza, że jest jedynym autorem tej pracy i zatwierdził ją do publikacji.
Oświadczenie o konflikcie interesów
Jestem dyrektorem ds. badań w Międzynarodowym Instytucie Konopi i Kannabinoidów. Obsługujemy klientów zaangażowanych w handel konopiami indyjskimi.
Podziękowania
Autor docenia pomoc pracowników University of Montana Inter-Library Loan Office of Mansfield Library w dostarczeniu materiałów badawczych.
Anderson, L. C. (1980). Leaf variation among Cannabis species from a controlled garden. Bot. Mus. Lealf. Harv. Univ. 28, 61-69.
Google Scholar
Appendino, G., Taglialatela-Scafati, O., Minassi, A., Pollastro, F., Ballero, L. M., Maxia, A., et al. (2015). Helichrysum italicum: śpiący olbrzym śródziemnomorskiego ziołolecznictwa. Herbalgram 105, 34-45.
Google Scholar
Bailey, L. H., and Bailey, E. Z. (1976). Hortus Third: A Concise Dictionary of Plants Cultivated in the United States and Canada. New York, NY: Macmillan.
Google Scholar
Ben-Shabat, S., Fride, E., Sheskin, T., Tamiri, T., Rhee, M. H., Vogel, Z., et al. (1998). An entourage effect: inactive endogenous fatty acid glycerol esters enhance 2-arachidonoyl-glycerol cannabinoid activity. Eur. J. Pharmacol. 353, 23-31. doi: 10.1016/S0014-2999(98)00392-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Berman, P., Futoran, K., Lewitus, G. M., Mukha, D., Benami, M., Shlomi, T., et al. (2018). A new ESI-LC/MS approach for comprehensive metabolic profiling of phytocannabinoids in Cannabis. Sci. Rep. 8:14280. doi: 10.1038/s41598-018-32651-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Blasco-Benito, S., Seijo-Vila, M., Caro-Villalobos, M., Tundidor, I., Andradas, C., Garcia-Taboada, E., et al. (2018). Ocena „efektu świty”: działanie przeciwnowotworowe czystego kannabinoidu w porównaniu z botanicznym preparatem leczniczym w przedklinicznych modelach raka piersi. Biochem. Pharmacol. 157, 285-293. doi: 10.1016/j.bcp.2018.06.025
PubMed Abstract | CrossRef Full Text | Google Scholar
Bohlmann, F., and Hoffmann, E. (1979). Cannabigerol-ahnliche verbindungen aus Helichrysum umbraculigerum. Phytochemistry 18, 1371-1374. doi: 10.1016/0031-9422(79)83025-3
CrossRef Full Text | Google Scholar
Bonn-Miller, M. O., ElSohly, M. A., Loflin, M. J. E., Chandra, S., and Vandrey, R. (2018). Rozwój leków z konopi indyjskich i kannabinoidów: ocena podejść botanicznych versus jednocząsteczkowych. Int. Rev. Psychiatry 30, 277-284. doi: 10.1080/09540261.2018.1474730
PubMed Abstract | CrossRef Full Text | Google Scholar
Booth, J. K., Page, J. E., and Bohlmann, J. (2017). Terpene synthases from Cannabis sativa. PLoS One 12:e0173911. doi: 10.1371/journal.pone.0173911
PubMed Abstract | CrossRef Full Text | Google Scholar
Brickell, C. D., Alexander, C., David, J. C., Hetterscheid, W. L. A., Leslie, A. C., Malecot, V., et al. (2009). International Code of Nomenclature for Cultivated Plants (Międzynarodowy Kodeks Nomenklatury Roślin Uprawnych). Leuven: International Society for Horticultural Science.
Google Scholar
Carvalho, A., Hansen, E. H., Kayser, O., Carlsen, S., and Stehle, F. (2017). Designing microorganisms for heterologous biosynthesis of cannabinoids. FEMS Yeast Res. 17:fox037. doi: 10.1093/femsyr/fox037
PubMed Abstract | CrossRef Full Text | Google Scholar
Clarke, R. C., and Merlin, M. D. (2013). Cannabis: Evolution and Ethnobotany. Berkeley, CA: University of California Press.
Google Scholar
Clarke, R. C., and Merlin, M. D. (2016). Udomowienie Cannabis, historia hodowli, obecna różnorodność genetyczna i perspektywy na przyszłość. Crit. Rev. Plant Sci. 35, 293-327. doi: 10.1080/07352689.2016.1267498
CrossRef Full Text | Google Scholar
Cunha, J. M., Carlini, E. A., Pereira, A. E., Ramos, O. L., Pimentel, C., Gagliardi, R., et al. (1980). Chronic administration of cannabidiol to healthy volunteers and epileptic patients. Pharmacology 21, 175-185. doi: 10.1159/000137430
PubMed Abstract | CrossRef Full Text | Google Scholar
Datwyler, S. L., and Weiblen, G. D. (2006). Genetic variation in hemp and marijuana (Cannabis sativa L.) according to amplified fragment length polymorphisms. J. Forensic Sci. 51, 371-375. doi: 10.1111/j.1556-4029.2006.00061.x
PubMed Abstract | CrossRef Full Text | Google Scholar
de Meijer, E. (2004). „The breeding of cannabis cultivars for pharmaceutical end uses,” in Medicinal Uses of Cannabis and Cannabinoids, eds G. W. Guy, B. A. Whittle, and P. Robson (London: Pharmaceutical Press), 55-70.
Google Scholar
de Meijer, E. P., Bagatta, M., Carboni, A., Crucitti, P., Moliterni, V. M., Ranalli, P., et al. (2003). The inheritance of chemical phenotype in Cannabis sativa L. Genetics 163, 335-346.
PubMed Abstract | Google Scholar
de Meijer, E. P. M., and Hammond, K. M. (2005). The inheritance of chemical phenotype in Cannabis sativa L. (II): cannabigerol predominant plants. Euphytica 145, 189-198. doi: 10.1007/s10681-005-1164-8
CrossRef Full Text | Google Scholar
de Meijer, E. P. M., Hammond, K. M., and Micheler, M. (2009a). The inheritance of chemical phenotype in Cannabis sativa L. (III): variation in cannabichromene proportion. Euphytica 165, 293-311.
Google Scholar
de Meijer, E. P. M., Hammond, K. M., and Sutton, A. (2009b). The inheritance of chemical phenotype in Cannabis sativa L. (IV): cannabinoid-free plants. Euphytica 168, 95-112.
Google Scholar
Devinsky, O., Cross, J. H., Laux, L., Marsh, E., Miller, I., Nabbout, R., et al. (2017). Trial of cannabidiol for drug-resistant seizures in the dravet syndrome. N. Engl. J. Med. 376, 2011-2020. doi: 10.1056/NEJMoa1611618
PubMed Abstract | CrossRef Full Text | Google Scholar
Devinsky, O., Marsh, E., Friedman, D., Thiele, E., Laux, L., Sullivan, J., et al. (2016). Cannabidiol u pacjentów z padaczką oporną na leczenie: otwarte badanie interwencyjne. Lancet Neurol. 15, 270-278. doi: 10.1016/S1474-4422(15)00379-8
CrossRef Full Text | Google Scholar
Devinsky, O., Patel, A. D., Thiele, E. A., Wong, M. H., Appleton, R., Harden, C. L., et al. (2018). Randomizowane, zróżnicowane pod względem dawki badanie bezpieczeństwa kannabidiolu w zespole Dravet. Neurology 90, e1204-e1211. doi: 10.1212/WNL.000000005254
PubMed Abstract | CrossRef Full Text | Google Scholar
Dufresnes, C., Jan, C., Bienert, F., Goudet, J., and Fumagalli, L. (2017). Różnorodność genetyczna Cannabis na szeroką skalę dla zastosowań kryminalistycznych. PLoS One 12:e0170522. doi: 10.1371/journal.pone.0170522
PubMed Abstract | CrossRef Full Text | Google Scholar
Elzinga, S., Fischedick, J., Podkolinski, R., and Raber, J. C. (2015). Kannabinoidy i terpeny jako markery chemotaksonomiczne w konopiach indyjskich. Nat. Prod. Chem. Res. 3:181.
Google Scholar
Farhi, M., Marhevka, E., Masci, T., Marcos, E., Eyal, Y., Ovadis, M., et al. (2011). Harnessing yeast subcellular compartments for the production of plant terpenoids. Metab. Eng. 13, 474-481. doi: 10.1016/j.ymben.2011.05.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Food and Drug Administration (2015). Botanical Drug Development Guidance for Industry, wyd. U.S.D.o.H.a.H. Services (Washington, DC: Food and Drug Administration). doi: 10.1016/j.ymben.2011.05.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Fuchs, L. (1999). The Great Herbal of Leonhart Fuchs: De Historia Stirpium Commentarii Insignes, 1542 (Notable Commentaries on the History of Plants). Stanford, CA: Stanford University Press.
Google Scholar
Gallily, R., Yekhtin, Z., and Hanus, L. (2014). Overcoming the bell-shaped dose-response of cannabidiol by using cannabis extract enriched in cannabidiol. Pharmacol. Pharm. 6, 75-85. doi: 10.4236/pp.2015.62010
CrossRef Full Text | Google Scholar
Gaoni, Y., and Mechoulam, R. (1964). Isolation, structure and partial synthesis of an active constituent of hashish. J. Am. Chem. Soc. 86, 1646-1647. doi: 10.1021/ja01062a046
CrossRef Full Text | Google Scholar
Goldstein, B. (2016). Cannabis w leczeniu padaczki dziecięcej. Chicago, IL: O’Shaughnessy’s, 7-9.
Google Scholar
Hillig, K. W. (2005a). A combined analysis of agronomic traits and allozyme allele frequencies for 69 Cannabis accessions. J. Ind. Hemp 10, 17-30. doi: 10.1300/J237v10n01_03
CrossRef Full Text | Google Scholar
Hillig, K. W. (2005b). Genetic evidence for speciation in Cannabis (Cannabaceae). Genet. Resour. Crop Evol. 52, 161-180. doi: 10.1007/s10722-003-4452-y
PubMed Abstract | CrossRef Full Text | Google Scholar
Hillig, K. W., and Mahlberg, P. G. (2004). A chemotaxonomic analysis of cannabinoid variation in Cannabis (Cannabaceae). Am. J. Bot. 91, 966-975. doi: 10.3732/ajb.91.6.966
PubMed Abstract | CrossRef Full Text | Google Scholar
Johnson, J. R., Burnell-Nugent, M., Lossignol, D., Ganae-Motan, E. D., Potts, R., and Fallon, M. T. (2010). Wieloośrodkowe, podwójnie ślepe, randomizowane, kontrolowane placebo, równoległe badanie skuteczności, bezpieczeństwa i tolerancji ekstraktu THC:CBD i ekstraktu THC u pacjentów z trudnym do opanowania bólem nowotworowym. J. Pain Symptom Manage. 39, 167-179. doi: 10.1016/j.jpainsymman.2009.06.008
PubMed Abstract | CrossRef Full Text | Google Scholar
Lamarck, J. B. (1783). Encyclopédie Méthodique Botanique. Paris: Chez Panckoucke.
Lewis, M. A., Russo, E. B., and Smith, K. M. (2018). Farmakologiczne podstawy chemowarów Cannabis. Planta Med. 84, 225-233. doi: 10.1055/s-0043-122240
PubMed Abstract | CrossRef Full Text | Google Scholar
Linnaeus, C. (1753). Species Plantarum. Holmiae: Laurentii Salvii.
Google Scholar
McPartland, J., Guy, G. W., and Hegman, W. (2018). Konopie indyjskie są rdzenne dla Europy, a uprawa rozpoczęła się w epoce miedzi lub brązu: probabilistyczna synteza badań kopalnych pyłków. Veg. His. Archaeobot. 27, 635-648. doi: 10.1007/s00334-018-0678-7
CrossRef Full Text | Google Scholar
McPartland, J. M. (2018). Systematyka Cannabis na poziomie rodziny, rodzaju i gatunku. Cannabis Cannabinoid Res. 3, 203-212. doi: 10.1089/can.2018.0039
PubMed Abstract | CrossRef Full Text | Google Scholar
McPartland, J. M., Clarke, R. C., and Watson, D. P. (2000). Hemp Diseases and Pests: Management and Biological Control. Wallingford: CABI. doi: 10.1079/9780851994543.0000
CrossRef Full Text | Google Scholar
McPartland, J. M., and Guy, G. W. (2017). Modele taksonomii konopi, uprzedzenia kulturowe i konflikty między nazwami naukowymi i wernakularnymi. Bot. Rev. 83, 327-381. doi: 10.1007/s12229-017-9187-0
CrossRef Full Text | Google Scholar
McPartland, J. M., and Mediavilla, V. (2001). „Non-cannabinoids in cannabis,” in Cannabis and Cannabinoids, eds F. Grotenhermen and E. B. Russo (Binghamton, NY: Haworth Press), 401-409.
Google Scholar
McPartland, J. M., and Pruitt, P. L. (1999). Efekty uboczne farmaceutyków nie wywoływane przez porównywalne leki ziołowe: przypadek tetrahydrokannabinolu i marihuany. Altern. Ther. Health Med. 5, 57-62.
Google Scholar
McPartland, J. M., and Russo, E. B. (2001). Konopie indyjskie i ekstrakty z konopi: większe niż suma ich części? J. Cannabis Ther. 1, 103-132. doi: 10.1300/J175v01n03_08
CrossRef Full Text | Google Scholar
McPartland, J. M., and Russo, E. B. (2014). „Niefitokannabinoidowe składniki konopi i synergia ziołowa”, w Handbook of Cannabis, red. R. G. Pertwee (Oxford: Oxford University Press), 280-295.
Google Scholar
Mechoulam, R., and Ben-Shabat, S. (1999). From gan-zi-gun-nu to anandamide and 2-arachidonoylglycerol: the ongoing story of cannabis. Nat. Prod. Rep. 16, 131-143. doi: 10.1039/a703973e
PubMed Abstract | CrossRef Full Text | Google Scholar
Oswald, M., Fischer, M., Dirninger, N., and Karst, F. (2007). Monoterpenoid biosynthesis in Saccharomyces cerevisiae. FEMS Yeast Res. 7, 413-421. doi: 10.1111/j.1567-1364.2006.00172.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Pacher, P., and Mechoulam, R. (2011). Is lipid signaling through cannabinoid 2 receptors part of a protective system? Prog. Lipid Res. 50, 193-211. doi: 10.1016/j.plipres.2011.01.001
PubMed Abstract | CrossRef Full Text | Google Scholar
Pamplona, F. A., da Silva, L. R., and Coan, A. C. (2018). Potencjalne korzyści kliniczne ekstraktów Cannabis bogatych w CBD w stosunku do oczyszczonego CBD w padaczce opornej na leczenie: metaanaliza danych obserwacyjnych. Front. Neurol. 9:759. doi: 10.3389/fneur.2018.00759
PubMed Abstract | CrossRef Full Text | Google Scholar
Piomelli, D., and Russo, E. B. (2016). Debata Cannabis sativa versus Cannabis indica: wywiad z Ethanem Russo, MD. Cannabis Cannabinoid Res. 1, 44-46. doi: 10.1089/can.2015.29003.ebr
PubMed Abstract | CrossRef Full Text | Google Scholar
Russo, E. B. (2007). Historia konopi indyjskich i ich preparatów w sadze, nauce i sobowtórze. Chem. Biodivers. 4, 1614-1648. doi: 10.1002/cbdv.200790144
PubMed Abstract | CrossRef Full Text | Google Scholar
Russo, E. B. (2011). Taming THC: potential cannabis synergy and phytocannabinoid-terpenoid entourage effects. Br. J. Pharmacol. 163, 1344-1364. doi: 10.1111/j.1476-5381.2011.01238.x
PubMed Abstract | CrossRef Full Text | Google Scholar
Russo, E. B. (2016). Beyond Cannabis: rośliny i układ endokannabinoidowy. Trends Pharmacol. Sci. 37, 594-605. doi: 10.1016/j.tips.2016.04.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Russo, E. B. (2017). Cannabis i padaczka: starożytne leczenie powraca na pierwszy plan. Epilepsy Behav. 70(Pt B), 292-297. doi: 10.1016/j.yebeh.2016.09.040
PubMed Abstract | CrossRef Full Text | Google Scholar
Russo, E. B., Jiang, H. E., Li, X., Sutton, A., Carboni, A., del Bianco, F., et al. (2008). Phytochemical and genetic analyses of ancient cannabis from Central Asia. J. Exp. Bot. 59, 4171-4182. doi: 10.1093/jxb/ern260
PubMed Abstract | CrossRef Full Text | Google Scholar
Russo, E. B., and Marcu, J. (2017). Farmakologia konopi: zwykli podejrzani i kilka obiecujących tropów. Adv. Pharmacol. 80, 67-134. doi: 10.1016/bs.apha.2017.03.004
PubMed Abstract | CrossRef Full Text | Google Scholar
Russo, E. B., and McPartland, J. M. (2003). Cannabis to coś więcej niż tylko Delta(9)-tetrahydrokannabinol. Psychopharmacology 165, 431-432. doi: 10.1007/s00213-002-1348-z
PubMed Abstract | CrossRef Full Text | Google Scholar
Sawler, J., Stout, J. M., Gardner, K. M., Hudson, D., Vidmar, J., Butler, L., et al. (2015). Struktura genetyczna marihuany i konopi. PLoS One 10:e0133292. doi: 10.1371/journal.pone.0133292
PubMed Abstract | CrossRef Full Text | Google Scholar
Schultes, R., Klein, W., Plowman, T., and Lockwood, T. (1974). Cannabis: an example of taxonomic neglect. Bot. Mus. Lealf. Harv. Univ. 23, 337-367.
Google Scholar
Shoyama, Y., Takeuchi, A., Taura, F., Tamada, T., Adachi, M., Kuroki, R., et al. (2005). Crystallization of Delta1-tetrahydrocannabinolic acid (THCA) synthase from Cannabis sativa. Acta Crystallogr. Sect. F Struct. Biol. Cryst. Commun. 61(Pt 8), 799-801. doi: 10.1107/S1744309105023365
PubMed Abstract | CrossRef Full Text | Google Scholar
Sirikantaramas, S., Morimoto, S., Shoyama, Y., Ishikawa, Y., Wada, Y., Shoyama, Y., et al. (2004). The gene controlling marijuana psychoactivity: molecular cloning and heterologous expression of Delta1-tetrahydrocannabinolic acid synthase from Cannabis sativa L. J. Biol. Chem. 279, 39767-39774. doi: 10.1074/jbc.M403693200
PubMed Abstract | CrossRef Full Text | Google Scholar
Small, E. (2015). Ewolucja i klasyfikacja Cannabis sativa (marihuana, konopie) w odniesieniu do wykorzystania przez człowieka. Bot. Rev. 81, 189-294. doi: 10.1007/s12229-015-9157-3
CrossRef Full Text | Google Scholar
Small, E. (2017). Cannabis: A Complete Guide. Boca Raton, FL: CRC Press.
Google Scholar
Small, E., and Cronquist, A. (1976). A practical and natural taxonomy for Cannabis. Taxon 25, 405-435. doi: 10.2307/1220524
CrossRef Full Text | Google Scholar
Small, E., and Marcus, D. (2003). Poziomy tetrahydrokannabinolu w zasobach plazmy zarodkowej konopi (Cannabis sativa). Econ. Bot. 57, 545-558. doi: 10.1663/0013-0001(2003)0572.0.CO;2
CrossRef Full Text | Google Scholar
Song, B.-H., Wang, X.-Q., Li, F.-Z., and Hong, D.-Y. (2001). Furnter dowód dla the paraphyly the Celtidaceae od the chloroplast gene mat K. Plant Syst. Evol. 228, 107-115. doi: 10.1007/s006060170041
CrossRef Full Text | Google Scholar
Sulak, D., Saneto, R., and Goldstein, B. (2017). Obecny status marihuany rzemieślniczej w leczeniu padaczki w Stanach Zjednoczonych. Epilepsy Behav. 70(Pt B), 328-333. doi: 10.1016/j.yebeh.2016.12.032
PubMed Abstract | CrossRef Full Text | Google Scholar
Taura, F., Dono, E., Sirikantaramas, S., Yoshimura, K., Shoyama, Y., and Morimoto, S. (2007). Production of Delta(1)-tetrahydrocannabinolic acid by the biosynthetic enzyme secreted from transgenic Pichia pastoris. Biochem. Biophys. Res. Commun. 361, 675-680. doi: 10.1016/j.bbrc.2007.07.079
PubMed Abstract | CrossRef Full Text | Google Scholar
Taura, F., Morimoto, S., and Shoyama, Y. (1996). Purification and characterization of cannabidiolic-acid synthase from Cannabis sativa L. Biochemical analysis of a novel enzyme that catalyzes the oxidocyclization of cannabigerolic acid to cannabidiolic acid. J. Biol. Chem. 271, 17411-17416. doi: 10.1074/jbc.271.29.17411
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiele, E. A., Marsh, E. D., French, J. A., Mazurkiewicz-Beldzińska, M., Benbadis, S. R., Joshi, C., et al. (2018). Cannabidiol u pacjentów z napadami związanymi z zespołem Lennoxa-Gastauta (GWPCARE4): randomizowane, podwójnie zaślepione, kontrolowane placebo badanie fazy 3. Lancet 391, 1085-1096. doi: 10.1016/S0140-6736(18)30136-3
PubMed Abstract | CrossRef Full Text | Google Scholar
Usher, G. (1996). The Wordsmith Dictionary of Botany. Ware: Wordsworth Reference.
van Bakel, H., Stout, J. M., Cote, A. G., Tallon, C. M., Sharpe, A. G., Hughes, T. R., et al. (2011). The draft genome and transcriptome of Cannabis sativa. Genome Biol. 12:R102. doi: 10.1186/gb-2011-12-10-r102
PubMed Abstract | CrossRef Full Text | Google Scholar
Weigreffe, S. J., Sytsma, K. J., and Guries, R. P. (1998). The Ulmaceae, jedna rodzina czy dwie? Evidence from chloroplast DNA restriction site mapping. Plant Syst. Evol. 210, 249-270. doi: 10.1007/BF00985671
CrossRef Full Text | Google Scholar
Wilkinson, J. D., Whalley, B. J., Baker, D., Pryce, G., Constanti, A., Gibbons, S., et al. (2003). Medicinal cannabis: czy delta9-tetrahydrokannabinol jest niezbędny dla wszystkich efektów? J. Pharm. Pharmacol. 55, 1687-1694. doi: 10.1211/0022357022304
PubMed Abstract | CrossRef Full Text | Google Scholar
Wirtshafter, D. (1997). „Wartość odżywcza nasion konopi i oleju z nasion konopi”, w Cannabis w praktyce medycznej, red. M. L. Mathre (Jefferson, NC: McFarland and Company), 181-191.
Google Scholar
Xi, Z. X., Peng, X. Q., Li, X., Song, R., Zhang, H. Y., Liu, Q. R., et al. (2011). Brain cannabinoid CB(2) receptors modulate cocaine’s actions in mice. Nat. Neurosci. 14, 1160-1166. doi: 10.1038/nn.2874
PubMed Abstract | CrossRef Full Text | Google Scholar
Yang, M.-Q., van Velzen, R., Bakker, F. T., Sattarian, A., Li, D.-Z., and Yi, T.-S. (2013). Molecular phylogenetics and character evolution of Cannabaceae. Taxon 62, 473-485. doi: 10.12705/623.9
CrossRef Full Text | Google Scholar
Zirpel, B., Degenhardt, F., Martin, C., Kayser, O., and Stehle, F. (2017). Engineering yeasts as platform organisms for cannabinoid biosynthesis. J. Biotechnol. 259, 204-212. doi: 10.1016/j.jbiotec.2017.07.008
PubMed Abstract | CrossRef Full Text | Google Scholar
Zirpel, B., Degenhardt, F., Zammarelli, C., Wibberg, D., Kalinowski, J., Stehle, F., et al. (2018). Optimization of Delta(9)-tetrahydrocannabinolic acid synthase production in Komagataella phaffii via post-translational bottleneck identification. J. Biotechnol. 272-273, 40-47. doi: 10.1016/j.jbiotec.2018.03.008
PubMed Abstract | CrossRef Full Text
.