Il glicogeno e l’amido, polimeri di glucosio di dimensioni megadaltoniche, sono la principale riserva di energia e di composti di carbonio facilmente disponibili nella maggior parte degli organismi viventi. Nei mammiferi, l’assorbimento e l’utilizzo del glucosio sono sotto stretto controllo. Difetti nella normale manipolazione del glucosio sono associati a una varietà di patologie umane come le malattie da accumulo di glicogeno e il diabete, in cui l’iperglicemia persistente è correlata a un inizio precoce e a una maggiore gravità della malattia. Le vie metaboliche per la conversione catalitica del glicogeno coinvolgono diversi enzimi, tra questi la glicogeno sintasi catalizza l’allungamento e la glicogeno fosforilasi la scomposizione dei glucani a(1,4)-legati. Una grande ricchezza di informazioni è stata acquisita sulla degradazione del glicogeno attraverso gli studi biochimici e strutturali delle fosforilasi del glicogeno e della maltodestrina negli ultimi 30 anni. Questi studi hanno dimostrato che la regolazione della fosforilasi del glicogeno è un esempio affascinante di allosterismo covalente e non covalente, squisitamente coordinato dal controllo ormonale. Le informazioni che descrivono le strutture terziarie e quaternarie sono ora disponibili praticamente per tutti gli enzimi direttamente coinvolti nella trasformazione del glicogeno. Sorprendentemente, pochissime informazioni strutturali sono state riportate sulla glicogeno sintasi, che catalizza l’allungamento del glicogeno attraverso la successiva aggiunta di ![]() -(1,4)-residui di glucosio legati all’estremità non riducente del polimero, usando ADP/UDP-glucosio come donatore di glucosio.
-(1,4)-residui di glucosio legati all’estremità non riducente del polimero, usando ADP/UDP-glucosio come donatore di glucosio.
Abbiamo ora determinato la struttura cristallina della glicogeno sintasi di Agrobacterium tumefaciens (AtGS) con una risoluzione di 2,3 Å, sia nella sua forma apo che in presenza di ADP. I dati sono stati raccolti su ID29, e la struttura risolta usando la tecnica SAD (diffrazione anomala a lunghezza d’onda singola), seguita da procedure di modifica della densità. La proteina si trova come un dimero nell’unità asimmetrica, con ogni monomero composto da due domini “Rossmann-fold” (Figura 81) che sono organizzati come nelle strutture della glicogeno fosforilasi e altre glicosiltransferasi della superfamiglia GT-B. Una profonda fessura tra i due domini contiene il centro catalitico. Una grande differenza tra le glicogenosintasi dei mammiferi (famiglia GT3) e quelle batteriche (GT5) è che solo gli enzimi GT3 sono strettamente regolati dalla fosforilazione e dalla modulazione allosterica non covalente. Secondo gli allineamenti di sequenze multiple basati sulla struttura tra GT3, GT5 e sequenze di glicogeno fosforilasi, i siti di fosforilazione regolatori nella glicogeno sintasi umana sono probabilmente situati nelle inserzioni N-terminali di 25 residui e C-terminali di 120 residui, fuori dal nucleo GT-B non regolato delle glicogeno sintasi batteriche. Gli studi di mutagenesi avevano precedentemente identificato due cluster di Arg conservati negli enzimi GT3 come parte del sito/i allosterico/i sia per i residui fosfo-Ser/Thr che per l’attivatore non covalente Glc-6P. I residui equivalenti nell’AtGS si trovano a un’estremità del dominio C-terminale, lontano dal centro di reazione, indicando un effetto regolatore a lunga distanza.
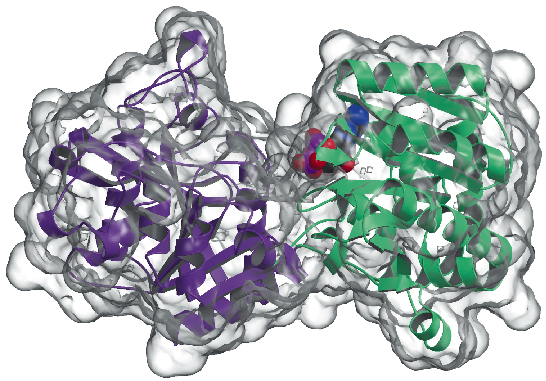
Fig. 81: Struttura della glicogeno sintasi: la superficie accessibile al solvente è rappresentata in trasparenza. Si noti la molecola di ADP legata (disegnata come palline VDW).
La struttura dell’AtGS mostra una sorprendente somiglianza topologica con le strutture centrali delle fosforilasi del glicogeno/maltodestrine (famiglia GT35) (Figura 82a), anche se le identità di sequenza sono basse (~14%). La struttura di ogni dominio Rossmann-fold di AtGS è significativamente più vicina a quelle delle fosforilasi della famiglia GT35 che a qualsiasi altra nucleotide-dipendente glicosil trasferasi. Le glicogeno sintasi e le fosforilasi mostrano anche una forte somiglianza nei loro siti catalitici (Figura 82b). Il ribosio ADP e i gruppi piridossali si trovano in posizioni equivalenti, così come il fosfato distale dell’ADP nelle AtGS e il substrato di fosfato inorganico nelle fosforilasi. Inoltre, anche gli amminoacidi critici che interagiscono con le società di glucosio dei substrati sono conservati. Il meccanismo catalitico usato dalle glicogeno sintasi e dalle fosforilasi è quindi probabilmente simile, con sottili cambiamenti nella struttura che spiegano le loro diverse proprietà funzionali complessive.
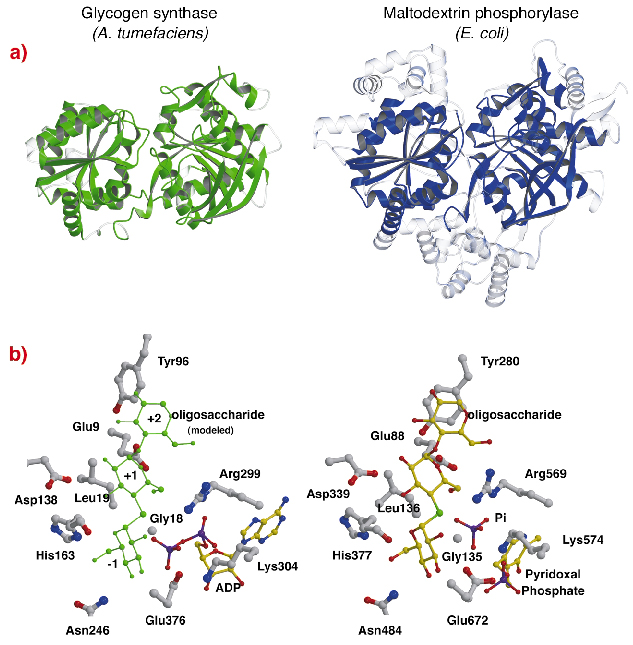
Fig. 82: Omologia alla glicogeno fosforilasi: (a) dopo l’allineamento strutturale, i residui equivalenti sovrapposti tra AtGS e la maltodestrina fosforilasi MalP di E. coli, sono rappresentati in materiale solido (regioni non allineate, in trasparente); (b) i siti catalitici di AtGS e MalP.
Il suggerimento iniziale che la sintesi del glicogeno rappresentasse semplicemente un’inversione della sua fosforolisi degradativa si è dimostrato errato quando Leloir ha dimostrato che l’UDP-Glc e la glicogeno sintasi, non la Glc-1-P e la glicogeno fosforilasi, erano responsabili dell’allungamento del polisaccaride. Il nostro lavoro dimostra ora, tuttavia, che le reazioni indipendenti opposte nell’interconversione glucosio-glicogeno sono catalizzate da enzimi omologhi strettamente correlati.