- Introduzione
- La base genetica della rottura degli ibridi nel riso
- Meccanismi molecolari alla base della rottura degli ibridi
- Rottura ibrida nell’allevamento convenzionale del riso
- Hybrid Breakdown in Genomics-Assited Breeding
- Discussione
- Data Availability Statement
- Author Contributions
- Conflitto di interessi
- Riconoscimenti
Introduzione
Gli allevatori e i ricercatori si sono interessati all’ibridazione e all’introgressione tra genotipi divergenti come potenziale motore che porta alla divergenza ecologica della progenie (Rieseberg et al., 2007; Arnold et al., 2012). Al contrario, l’ibridazione può talvolta comportare una ridotta fitness ibrida, come debolezza e/o sterilità nella F1 e nelle generazioni successive, anche nei casi di ibridazione tra membri della stessa specie.
La ridotta vitalità e/o fertilità ibrida che segrega nella F2 o nelle generazioni successive viene definita breakdown ibrido (HB), in cui gli alleli recessivi sono necessariamente associati. Questa barriera riproduttiva è stata osservata per molto tempo sia nelle piante che negli animali (Dobzhansky, 1970; Grant, 1971). Molte delle analisi genetiche di questa barriera hanno rivelato che è realizzata da un effetto complementare tra e/o tra loci con alleli differenziati, comunemente chiamato incompatibilità Bateson-Dobzhansky-Muller (BDM) (Rieseberg e Willis, 2007). L’HB implica necessariamente barriere riproduttive postzigotiche intrinseche, come l’inviabilità dell’ibrido (compresa la debolezza, la necrosi e la clorosi) e la sterilità ibrida (nel maschio, nella femmina o in entrambi i gameti); pertanto, alcuni ricercatori potrebbero non distinguere l’HB dall’inviabilità e dalla sterilità nella progenie F1. Tuttavia, ritengo che l’HB sia una classificazione conveniente per le barriere riproduttive perché implica la loro base genetica sottostante (cioè il coinvolgimento di alleli recessivi).
Studi recenti utilizzando la specie vegetale modello Arabidopsis hanno fornito una migliore comprensione dell’HB per quanto riguarda i suoi meccanismi molecolari (Vaid e Laitinen, 2019). Al contrario, sebbene il riso (specie Oryza) sia una coltura modello, l’attuale comprensione delle basi genetiche dell’HB rimane limitata in questa specie, probabilmente perché l’HB non è una barriera riproduttiva negli ibridi F1, in cui ci si aspetta una maggiore produzione di grano rispetto alle linee parentali inbred, e probabilmente perché le piante inferiori che segregano in F2 e nelle generazioni successive possono essere facilmente selezionate in base al fenotipo dalla popolazione di allevamento. La difficoltà di mappatura genetica causata dall’eredità recessiva può anche essere alla base di questa limitazione.
Qui, descrivo l’HB negli incroci di riso facendo riferimento alle informazioni fornite dagli studi sull’Arabidopsis, e discuto come l’HB viene gestita nel breeding del riso.
La base genetica della rottura degli ibridi nel riso
Si ritiene che il riso coltivato, O. sativa japonica e indica, formi un complesso di specie con il loro progenitore putativo (O. rufipogon) e le specie selvatiche; tuttavia, barriere riproduttive multiple, tra cui HB, sono osservate negli incroci tra loro (Oka, 1988; Vaughan et al, 2003).
Per quanto ne so, il Dr. Oka è stato il primo a descrivere la base genetica dell’HB negli incroci interspecifici tra japonica e indica, che era spiegata da due geni complementari (Oka, 1957). Tuttavia, i geni responsabili non sono stati mappati sui cromosomi, perché i marcatori molecolari non erano disponibili a quel tempo.
Negli ibridi di riso, HB è stato spesso descritto in incroci interspecifici (O. sativa ssp. japonica × ssp. indica) e interspecifici (O. sativa × O. nivara e O. sativa × O. glumaepatula) (Oka, 1957; Sato e Morishima, 1988; Wu et al, 1995; Li et al., 1997; Fukuoka et al., 1998, 2005; Kubo e Yoshimura, 2002, 2005; Matsubara et al., 2007a,b, 2015; Yamamoto et al., 2007; Ichitani et al., 2012;per incroci interspecifici; Sobrizal et al., 2001; Miura et al., 2008 per incroci interspecifici) (Tabella 1). La maggior parte di questi casi di HB sono stati causati da incompatibilità BDM a due loci e alleli recessivi, per cui 1/16 della progenie F2 che era omozigote per alleli recessivi ad entrambi i loci ha mostrato il fenotipo HB; tuttavia, 4/16 della progenie che era eterozigote ad un solo locus ha mostrato il fenotipo HB, a seconda della combinazione degli incroci (Tabella 1 e Figura 1A). Negli ibridi backcross, un locus era già fissato con gli alleli del genitore ricorrente; quindi, 1/4 della progenie BCnF2 ha mostrato il fenotipo HB (Tabella 1 e Figura 1B).

Tabella 1. Base genetica della ripartizione ibrida riportata negli incroci di riso.
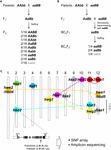
Figura 1. La base genetica della ripartizione ibrida (HB). (A) Rappresentazione schematica del genotipo HB che segrega nella progenie F2. Quando i genotipi parentali sono AAbb e aaBB, 1/16 della progenie F2 senza allele dominante, che è indicato dal carattere grigio (cioè, aabb), mostrano ridotta vitalità e/o fertilità, anche se gli altri genotipi sono normali. In alcuni incroci di riso, la progenie F2 con un solo allele dominante (cioè Aabb, aaBb) mostra anche una ridotta vitalità e/o fertilità. (B) Rappresentazione schematica del genotipo HB che segrega nella progenie BCnF2. Inoltre, 1/4 della progenie BCnF2 mostra una ridotta vitalità e/o fertilità. (C) Localizzazione cromosomica dei geni alla base di HB negli incroci di riso. I geni sono approssimativamente mappati in base ai risultati della mappatura dei geni in ogni studio. Una serie di geni complementari sono collegati dalla linea tratteggiata. I geni complementari con hbd1 e hwf1 non sono stati riportati. L’esempio del polimorfismo del nucleotide funzionale che causa HB pubblicato da Yamamoto et al. (2010) è mostrato, e queste informazioni ci permetteranno di condurre un’indagine tramite SNP array o amplicon sequencing.
Il fenotipo HB negli ibridi di riso varia in base alla combinazione di incroci. Per esempio, il genotipo hbd2/hbd2 hbd3/hbd3 mostra debolezza, ma nessuna sterilità dei semi evidente. Tuttavia, il genotipo hbd4/hbd4 hbd5/hbd5 mostra sia debolezza che sterilità dei semi (Matsubara et al., 2007a, 2015; Yamamoto et al., 2007; Tabella 1). Ci sono anche casi complicati in cui si osservano HB che mostrano sia debolezza che sterilità del seme (genotipo hwe1/hwe1 hwe2/hwe2) o HB che mostrano solo sterilità del seme (hsa1/hsa1 hsa2/hsa2 hsa3/hsa3), nonostante la stessa combinazione di incroci, cioè, Asominori × IR24 cross (Kubo e Yoshimura, 2002, 2005; Tabella 1). Poiché questi HB hanno basi genetiche indipendenti, almeno 5/64 della progenie F2 mostrerebbero il fenotipo HB in questo incrocio.
Ad oggi, diversi gruppi di loci responsabili dell’HB sono stati mappati in particolari regioni genomiche usando marcatori del DNA in incroci tra varietà di riso japonica e indica (Chrs 7 e 10, Fukuoka et al, 1998; Chrs 1 e 12, Wu et al., 1995; Kubo e Yoshimura, 2002; Matsubara et al., 2015; Chrs 6 e 11, Fukuoka et al., 2005; Chrs 8, 9, e 12, Kubo e Yoshimura, 2005; Chrs 2 e 11, Matsubara et al., 2007a; Yamamoto et al., 2007, 2010; Chrs 11 e 12, Ichitani et al., 2012; Tabella 1 e Figura 1C). I risultati di questi studi hanno rivelato che i loci alla base del riso HB sono condivisi in alcuni incroci, ma differiscono in altri incroci, il che è suggestivo della loro diversificazione nei genomi del riso. Per esempio, in alcuni casi l’allele HB portato da O. nivara, una specie strettamente legata alle varietà incica, condivide lo stesso locus con una varietà indica; tuttavia, l’allele HB portato da O. glumaepatula, una specie strettamente legata alle varietà coltivate, non condivide il locus con nessuna altra varietà (Tabella 1 e Figura 1C). Va notato che i casi di HB sopra descritti sono causati da un insieme di geni con effetti maggiori; tuttavia, ci sono anche casi causati da un insieme di geni con effetti minori, ad esempio una fertilità dei semi leggermente ridotta, che non può essere trascurata nel breeding del riso, che non sono ancora stati rilevati.
Meccanismi molecolari alla base della rottura degli ibridi
Risposta autoimmune: Bomblies et al. (2007) hanno riportato per primi che la risposta autoimmune che un gene NB-LRR di resistenza alle malattie o il gene R è associato all’HB dell’Arabidopsis negli incroci intraspecifici, anche se questa risposta autoimmune si esprime principalmente come necrosi nelle piante F1, e l’HB nella progenie F2 sembra essere condizionata dalla temperatura. Negli ibridi di Arabidopsis, sono state descritte diverse linee di evidenza di HB causata dalla risposta autoimmune (Alcázar et al., 2009, 2010). Il coinvolgimento dei geni NB-LRR nell’HB suggerisce che più regioni genomiche possono essere associate all’HB dell’Arabidopsis; infatti, l’ampia indagine condotta da Chae et al. (2014) supporta questa idea.
Anche negli ibridi di riso, l’HB causato da una risposta autoimmune è stato riportato in un incrocio japonica × indica (Yamamoto et al., 2010). In questo caso, l’HB si verifica in piante F2 quando il gene hybrid breakdown 2 (hbd2), che codifica per la caseina chinasi I ed è portato dalla varietà indica è combinato con un cluster di geni R portati dalla varietà japonica. Poiché è probabile che questi alleli causali si comportino come parzialmente recessivi, i prodotti del gene possono essere coinvolti (Matsubara et al., 2007a; Yamamoto et al., 2007, 2010).
Silenziamento reciproco di geni duplicati: questa nozione è stata proposta per la prima volta teoricamente come base genetica dell’incompatibilità ibrida (Werth e Windham, 1991; Lynch e Force, 2000). Nel modello, una popolazione perde la funzione in un locus e la conserva nell’altro, mentre l’altra popolazione sperimenta l’effetto opposto. Di conseguenza, 1/16 degli zigoti F2 non hanno geni funzionali (Lynch e Force, 2000). L’HB negli ibridi di Arabidopsis descritto da Bikard et al. (2009) e Vlad et al. (2010) è un buon esempio di questo fenomeno. In questo scenario, sono stati riportati anche casi che coinvolgono il silenziamento epigenetico (Agorio et al., 2017; Blevins et al., 2017).
Come il riso ha sperimentato sia la duplicazione dell’intero genoma che dei segmenti (Wang et al., 2007; Guo et al., 2019), il silenziamento reciproco potrebbe essere prevalente come causa di incompatibilità ibrida. Tuttavia, ad oggi, non ci sono prove di HB causata dal silenziamento reciproco di geni duplicati nel riso.
Indipendentemente dalla risposta autoimmune e dal silenziamento reciproco, è stata identificata un’interazione tra una DEAD-box RNA helicase 18 e un altro gene (MORPHEUS MOLECULE 1 come probabile candidato) che causa HB negli ibridi di Arabidopsis (Plötner et al., 2017; Vaid et al., 2020). È interessante notare che il fenotipo HB si riduce tra le generazioni F3 e F4, implicando una regolazione epigenetica dell’espressione genica.
Negli ibridi di riso, tra i tre loci di sterilità ibrida-a (hsa), Kubo et al. (2016) hanno recentemente dimostrato che il locus hsa1 consiste di due geni, e che questi geni codificano rispettivamente una proteina DUF1618 e una proteina non caratterizzata con qualche somiglianza con una proteina legante il nucleotide. Le caratteristiche molecolari dei restanti geni complementari, hsa2 e hsa3, non sono state riportate.
Rottura ibrida nell’allevamento convenzionale del riso
Per decenni, il metodo della popolazione di massa è stato ampiamente impiegato nell’allevamento convenzionale del riso. In questo metodo, dopo l’incrocio, la popolazione F4 o F5 viene allevata per autofecondazione alla rinfusa (piuttosto che con il metodo di discesa a seme singolo) senza selezione artificiale, ma la popolazione della prima generazione è sottoposta a selezione naturale e di vitalità (Allard, 1960; Ikehashi e Fujimaki, 1980 per i dettagli). Nel metodo della bulk-population, ci si può aspettare che molti genotipi deboli e/o sterili vengano eliminati dalla popolazione prima della creazione di una popolazione di generazione avanzata; pertanto, questo metodo può fornire informazioni limitate sulla HB.
Per l’introgressione o l’accumulo di tratti desiderabili da donatore/i a una varietà, i selezionatori di riso hanno spesso eseguito il backcrossing o l’incrocio di più genitori, seguito dal metodo della bulk-population. Empiricamente, è noto da tempo che questi metodi di incrocio permettono di mitigare la perdita di candidati alla selezione a causa delle barriere riproduttive, perché questi metodi spesso riducono la segregazione delle combinazioni di alleli dirompenti associate alla HB in una progenie ibrida. Tuttavia, l’attenuazione delle barriere riproduttive con questi metodi di incrocio dipende inevitabilmente dal genotipo HB dei genitori.
Hybrid Breakdown in Genomics-Assited Breeding
La pubblicazione delle sequenze del genoma delle colture di riferimento e lo sviluppo delle tecnologie di sequenziamento di prossima generazione hanno accelerato il progresso dell’allevamento molecolare delle colture (Kole et al., 2015). In questo contesto, l’allevamento assistito dalla genomica, come la selezione genomica basata su genotipi di marcatori del DNA a livello di genoma, è stato considerato nell’allevamento delle colture (Spindel e Iwata, 2018).
Nell’allevamento assistito dalla genomica delle colture autoimpollinate, le popolazioni di generazione avanzata, come le linee inbred ricombinanti, sono spesso utilizzate come popolazioni di riferimento, dalle quali si ottengono dati sul genotipo e sul fenotipo a livello di genoma. Successivamente, la selezione basata solo sui genotipi dei marcatori viene effettuata nella progenie delle prime generazioni (ad esempio, F2). In questo schema di selezione, va notato che la popolazione di riferimento di solito non fornisce le informazioni della regione genomica per HB, perché la progenie debole e/o sterile dovrebbe essere stata eliminata nelle prime generazioni dopo l’incrocio. Pertanto, se tale schema di selezione viene adottato, dovremo abbandonare alcuni importanti candidati alla selezione. In alternativa, potremmo selezionare candidati indesiderabili in modo che l’HB diventi evidente nelle generazioni successive.
Discussione
Nonostante il numero limitato di studi su questo argomento, sembra che i loci alla base dell’HB negli incroci di riso siano diversificati piuttosto che condivisi (Tabella 1 e Figura 1). Come descritto sopra, le informazioni sulla distribuzione degli alleli associati all’HB tra i genitori dell’incrocio dovrebbero essere un prerequisito per il breeding del riso, in particolare per massimizzare l’efficacia del breeding assistito dalla genomica. Pertanto, è necessario rilevare ulteriormente l’HB negli incroci di riso e la mappatura dei geni responsabili nel genoma del riso. Negli ibridi di Arabidopsis, tali dati sui geni R e sui loro geni interagenti sono stati ampiamente studiati (Alcázar et al., 2010, 2014; Chae et al., 2014). La segnalazione del gene hbd2 da Yamamoto et al. (2010) in un ibrido di riso è un buon esempio di una vasta indagine eseguita utilizzando gli array SNP o il sequenziamento amplicone (Figura 1C). Anche i geni hbd3 e hsa1 sono candidati per questo tipo di indagine. Alla fine, lo sviluppo di criteri che ci permettono di predire l’HB sulla base delle informazioni genomiche delle linee parentali servirà come strumento importante per l’allevamento assistito dalla genomica. Anche se un fattore causale non è stato identificato come un singolo gene, i marcatori strettamente collegati, come i polimorfismi a singolo nucleotide, saranno efficaci per classificare gli aplotipi associati all’HB, perché il linkage disequilibrium del riso coltivato è stato stimato in 100-200 kb, anche se quello delle specie selvatiche (come O. rufipogon) può estendersi su diverse decine di kb (Mather et al., 2007; McNally et al., 2009; Huang et al., 2010). Queste informazioni permetteranno di progettare una combinazione di incroci più efficiente ed efficace.
L’analisi dei segreganti seguiti dal sequenziamento di prossima generazione può essere utile per la mappatura dei loci associati all’HB, così come per la mappatura di linkage convenzionale (come l’analisi dei locus dei tratti quantitativi), perché i segreganti HB spesso mostrano caratteristiche distinguibili (su tratti come l’altezza della pianta, il numero di tiller e la fertilità) dai segreganti di crescita normale in ogni popolazione di incrocio. L’acquisizione di dati di imaging utilizzando una fotocamera digitale e il caricamento di un drone può anche svolgere un ruolo importante nella fenotipizzazione in laboratorio e in risaia. Questi sforzi forniranno informazioni preziose non solo ai selezionatori di riso, ma anche ai biologi evolutivi.
Inoltre, se viene identificato un allele associato a HB, l’interruzione del gene attraverso il fascio di ioni o l’editing del genoma può aiutare a superare HB, come esemplificato dalla sterilità ibrida F1 nel riso (Koide et al., 2018; Xie et al, 2019).
Data Availability Statement
Tutti i dataset presentati in questo studio sono inclusi nell’articolo/materiale supplementare.
Author Contributions
L’autore conferma di essere l’unico contributore di questo lavoro e lo ha approvato per la pubblicazione.
Conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Riconoscimenti
Vorrei dedicare questo articolo alla memoria del Dr. Darshan Brar. Era un eminente selezionatore di riso che ha vigorosamente introdotto geni utili portati da specie selvatiche in cultivar al di là delle barriere riproduttive.
Allard, R. W. (1960). Principles of Plant Breeding. New York, NY: John Wily and Sons, Inc.
Google Scholar
Dobzhansky, T. (1970). Genetica del processo evolutivo. New York, NY: Columbia University Press.
Google Scholar
Grant, V. (1971). Speciazione delle piante. New York, NY: Columbia University Press.
Google Scholar
Ikehashi, H., and Fujimaki, H. (1980). “Modified bulk population method for rice breeding”, in Proceedings of the International Rice Research Conference on Innovative approaches to rice breeding, (Los Baños, PA: International Rice Research Institute), 163-182.
Google Scholar
Oka, H.-I. (1988). Origine del riso coltivato. Tokyo: JSSP.
Google Scholar